Lexikon der Biologie: Replikation
Replikationw [von *replik- ; Verb replizieren, Adj. replikativ], Reduplikation, Autoreduplikation, die identische Verdopplung oder Vervielfachung von DNA (bzw. von RNA bei den RNA-Viren). Die Replikation ist die molekulare Grundlage für die Weitergabe der genetischen Information von Generation zu Generation. Mit Ausnahme bestimmter Viren, bei denen einzelsträngige DNA (einzelsträngige DNA-Phagen) bzw. RNA (einzelsträngige RNA-Phagen) repliziert wird, erfolgt die Replikation an doppelsträngiger DNA (Doppelstrang) nach dem semikonservativen Modus, d.h., die beiden durch bidirektionale Verlängerung bei einer Replikationsrunde entstehenden sog. Tochter-DNA-Moleküle enthalten je einen elterlichen und einen neusynthetisierten Einzelstrang. Diese Verlängerung erfolgt von einem einzigen Startpunkt aus ( vgl. Abb. 1 ). Der Gesamtprozeß unterteilt sich in die 3 Hauptphasen Initiation, Elongation und Termination, wovon jedoch nur die beiden ersten Phasen, besonders durch Mutantenanalyse und in-vitro-Systeme, gut untersucht sind. Zahlreiche Einzelschritte der Replikation konnten besonders an Bakteriophagen-DNAs (Bakteriophagen) als Modellsystemen und an bakterieller DNA (Bakterienchromosom) untersucht werden. Die Enzyme und anderen Proteinfaktoren, die z.B. bei Escherichia coli und eukaryotischen Organismen die Replikation durchführen, sind zueinander analog. Mit einzelsträngiger Bakteriophagen-DNA, deren Replikation jedoch in mehreren Aspekten (z.B. Wechsel einzel- und doppelsträngiger Zwischenstufen) atypisch ist (rolling circle-Prinzip), gelang 1967 sogar die Replikation biologisch aktiver DNA im Reagenzglas ( Replikation der DNA I ). In bakteriellen Systemen, besonders in E.coli, konnten zahlreiche Enzymproteine (u.a. DNA-Polymerasen, als Primasen wirkende RNA-Polymerasen, DNA-Ligase, DNA-Helicasen, DNA-Topoisomerasen, Exonucleasen) und die von diesen katalysierten Einzelschritte der Replikation charakterisiert werden. – Die Proteine sind teilweise an bzw. in der Nähe der sog. Replikationsgabel ( vgl. Abb. 2 und vgl. Abb. 3 ) zu einem als Replisom bezeichneten Komplex vereinigt, in dem u.a. DNA-Polymerasen und Primasen enthalten sind. Letztere katalysieren die für die Replikation erforderliche primer-RNA-Synthese und sind zusammen mit anderen Proteinen in einem sog. Primosom-Komplex (Primosom, DNA-Primase) vereinigt. Neuere Untersuchungen deuten darauf hin, daß die Polymerase bei der Replikation nicht, wie bislang angenommen, am Matrizenstrang entlangfährt, sondern daß das Enzym an bestimmten Bereichen im Zellkern fixiert ist und die DNA mittels eines noch nicht bekannten Mechanismus durch die Polymerase hindurchgezogen wird. Während der DNA-Synthese katalysieren DNA-Polymerasen die Verlängerung eines Starterfragments zum vollständigen komplementären (Komplementarität) Tochter-DNA-Strang gegenüber einer elterlichen DNA-Matrize (engl. template). DNA-Polymerasen können DNA generell nur in 5' -› 3'-Richtung synthetisieren, weshalb bei der Replikation nur einer der beiden neusynthetisierten DNA-Stränge unmittelbar durch „Vorwärts-Replikation“ als fortlaufende Kette (der in 5' -› 3'-Richtung wachsende, „führende“ Strang, engl. leading strand; auch als Leitstrang bezeichnet) entstehen kann, während der andere Strang zunächst in Form kürzerer, ca. 1000 Nucleotide enthaltender Ketten (nach dem Entdecker Okazaki-Fragmente genannt) aufgebaut wird, die erst durch Folgeschritte (Beseitigung von RNA-primern, Auffüllen der kleinen Einzelstranglücken; Ligation) zu einem durchgehenden Strang umgewandelt werden. Für den aufgrund dieser „Rückwärts-Replikation“ verzögert entstehenden DNA-Strang wurde die engl. Bezeichnung lagging strand („verzögerter“ Strang; auch als Folgestrang bezeichnet) eingeführt. Diese Form der Replikation, kontinuierlich am Leitstrang und diskontinuierlich am Folgestrang, wird als semidiskontinuierlich bezeichnet. – Die Replikation startet an einer oder mehreren definierten Stellen der zu replizierenden DNA, die Replikationsursprung (origin of replication; ori) genannt werden. Die Replikationsursprünge einfacher Systeme (virale DNAs, Plasmide, bakterielle DNAs) konnten lokalisiert und in der Nucleotidsequenz aufgeklärt werden. Z.B. umfaßt der E.coli-Replikationsursprung oriC etwa 245 Basenpaare (bp) und weist 9-bp-Sequenzwiederholungen (repeats) und AT-reiche 13-bp-Sequenzen auf, die man auch als 9er- bzw. 13er-Sequenzen bezeichnet. An die oriC-Region grenzt ein DNA-Abschnitt mit hohem AT-Anteil, was die Entwindung (Rep-Protein) des DNA-Doppelstrangs erleichtert, da für das Aufschmelzen (schmelzen) von A/T-Basenpaaren weniger Energie erforderlich ist als für G/C-Basenpaare. Die genannten einfacheren bakteriellen Systeme weisen in der Regel nur 1 Replikationsursprung auf, jeder DNA-Ring repräsentiert 1 Replikationseinheit, ein Replikon. Bakteriengenome sind somit monoreplikonisch. Im Gegensatz dazu wird die Replikation chromosomaler DNA höherer Zellen zum Teil gleichzeitig an mehr als 1000 Stellen initiiert, eukaryotische Chromosomen sind multireplikonisch. Die Replikation in Zellen Höherer Organismen findet in der S-Phase (8–12 h) des Zellzyklus ( Mitose ) statt. Bei sehr kurzen S-Phasen (10–15 min), wie z.B. während des Furchungsstadiums (Furchung) in der Embryonalentwicklung, kann die Replikation sowohl durch erhöhte Synchronisation der bereits aktiven Replikationsursprünge als auch durch Aktivierung zusätzlicher Replikationsursprünge beschleunigt und damit der Zellzyklusgeschwindigkeit angepaßt werden. Das menschliche Genom enthält wahrscheinlich 10.000 bis 100.000 Replikons, die jedoch nicht während der gesamten Replikationsdauer aktiv sind. Die Vielzahl der Oris bedingt eine genaue Kontrolle, da in jedem Zellzyklus nur einmal repliziert werden darf. Für diese Kontrolle sind 2 Proteinkomplexe zuständig, der orc oder ORC (origin recognition complex) und der rlf oder RLF (replication licensing factor), die wiederum von CDC-Proteinen (cell division cycle; cdc-Gene) und Cyclin-abhängigen Proteinkinasen kontrolliert werden ( vgl. Infobox ). – Als Ansatzstellen für die Replikation konnten in neuerer Zeit als CpG-Inseln bezeichnete Bereiche identifiziert werden, die durch viele nicht methylierte Zweiergruppen von Cytosin und Guanin auffallen. Der Replikationskomplex kann sich offenbar nur dann an der DNA anlagern, wenn keine Methylgruppen im Wege sind. Derartige Sequenzauffälligkeiten konnten auch für die Startstellen der Transkription nachgewiesen werden, was auf einen Zusammenhang zwischen der Genaktivität (Transkription) und der Replikation hindeutet. Als selektive Replikation einzelner Gene ist die ebenfalls bei Höheren Organismen beobachtete Genamplifikation zu nennen. Im Vergleich zu ringförmigen Bakterien-Genomen ist die Replikation linearer chromosomaler DNA in einem weiteren Aspekt unterschiedlich. Die eukaryotischen Chromosomen besitzen spezielle Enden, die Telomere. Nähert sich die Replikationsgabel dem Ende des linearen Chromosoms, so wird der Leitstrang bis zum Ende der DNA-Matrize synthetisiert und der komplette Tochterstrang freigesetzt. Der Folgestrang wird jedoch diskontinuierlich kopiert und kann daher nicht vollständig bis zum Ende repliziert werden. Der Folgestrang würde also bei jeder Replikation kürzer werden. Diese ständige Verkürzung wird durch ein Enzym, die sog. Telomerase, verhindert, die an das 3'-Ende der Folgestrangmatrize Nucleotide anhängt. Nach erfolgter Replikation werden die Tochter-Helices wieder mit Histonen komplexiert. Die Histone, die bereits vor der Replikation an das Chromosom gebunden waren, finden sich nach der Replikation an dem DNA-Strang, der den Leitstrang enthält. Die Verteilung der Histone ist also konservativ, im Gegensatz zur semikonservativen DNA-Replikation. – Durch in vitro-Versuche konnte gezeigt werden, daß die Häufigkeiten von Einbaufehlern durch DNA-Polymerasen zwischen 10–4 und 10–5 liegen. Die durch Einbaufehler (Basenanaloga) entstehenden Fehlpaarungen zwischen elterlichem und neusynthetisiertem DNA-Strang können jedoch durch Reparaturprozesse (DNA-Reparatur), besonders durch die in DNA-Polymerasen häufig integrierte 3'-5'-Exonuclease-Aktivität, weitgehend beseitigt werden, wodurch Gesamtfehlerraten von < 10–8 pro Polymerisationsschritt resultieren. – Die Replikationsgeschwindigkeit des Bakterienchromosoms beträgt bei E.coli etwa 500–1000 bp/s, so daß die Replikation mehr Zeit in Anspruch nimmt, als die unter optimalen Wachstumsbedingungen alle 20 min erfolgende Teilung (mikrobielles Wachstum). Aus diesem Grund lassen sich mehrere Replikationsgabeln beobachten. Bei Eukaryoten erfolgt die Replikation wesentlich langsamer, wobei Werte unter 100 bp/s bestimmt wurden. Desoxyribonucleinsäuren, dispersive Replikation, displacement loop, Endoreplikation, Leben; Replikation der DNA IReplikation der DNA II .
H.K./S.Kl.
Lit.:Kornberg, A., Baker, T.: DNA replication. New York 21992.
Replikation
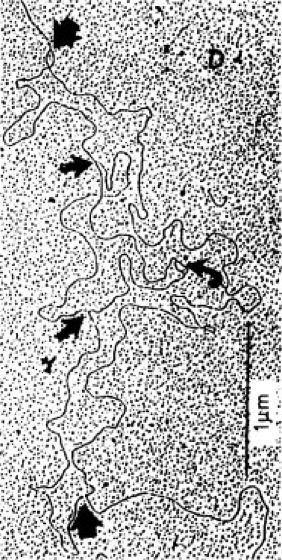
Abb. 1: Elektronenmikroskopisches Bild einer bidirektionalen DNA-Replikation. Die Replikation der meisten DNA-Moleküle ist bidirektional, d.h., sie verläuft von einem Startpunkt aus in beide Richtungen. Die DNA der Viren und der Bakterien besitzt nur einen einzigen Anfangspunkt für die Replikation. Dieser wird für eine zweite Replikationsrunde wiederbenutzt, bevor die erste Replikationsrunde ganz durchgelaufen ist. In der Abb. sind 3 laufende Replikationsrunden zu erkennen, die alle am selben Startpunkt begonnen haben, aber zeitlich versetzt, so daß sie verschieden weit fortgeschritten sind.
Replikation
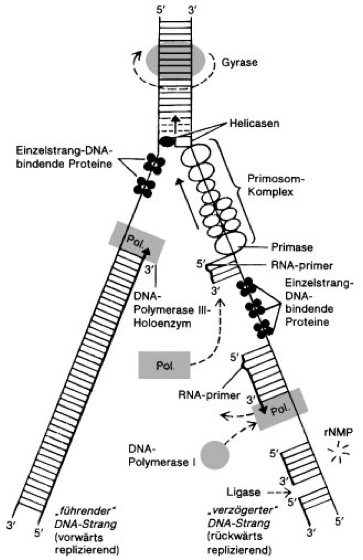
Abb. 2:Replikationsgabel (Modell für E.coli) mit der Vorwärts-Replikation des einen und der – stückweisen – Rückwärts-Replikation des anderen Tochterstrangs sowie Übersicht der an der DNA-Replikation beteiligten Enzymproteine und Reaktionsschritte. DNA-Stränge sind durch gerade, RNA-Stränge durch gewellte Linien dargestellt; die Windung der DNA-Helices ist daher nicht berücksichtigt. Nach der lokalen Entwindung der DNA am Replikationsursprung (Ori, ori) wird die Doppelhelix durch einzelstrangspezifische DNA-abhängige Helicasen weiter entwunden. Die weitere Stabilisierung erfolgt durch die kooperative Bindung des SSB-Proteins (Einzelstrang-Bindungsprotein, single strand binding protein; Abk. ssBP). Auftretende Torsionsspannungen werden von Topoisomerasen, bei E.coli von der Gyrase, aufgehoben. Die Pfeilspitzen kennzeichnen die arbeitenden DNA-Polymerase-III-Moleküle und weisen entsprechend der Syntheserichtung von 5' nach 3'. Am Leitstrang erfolgt eine kontinuierliche Synthese des neuen Strangs, am Folgestrang arbeitet die DNA-Polymerase III dagegen rückwärts und verlängert die 3'-Enden von RNA-primern. Die nach der Synthese durch DNA-Polymerase III (Pol.) verbleibenden Lücken des verzögert synthetisierten DNA-Strangs werden durch die langsamer arbeitende DNA-Polymerase I aufgefüllt, wobei gleichzeitig die RNA-primer durch die 5'-3'-Exonuclease-Aktivität von DNA-Polymerase bzw. durch eine RNA-DNA-Hybrid-spezifische RNase (sog. RNase H) unter Bildung von Ribonucleosidmonophosphaten (rNMP) abgebaut werden. Die aufgrund dieser Reaktionen jetzt nahtlos nebeneinander passenden, jedoch noch durch 3 Strangbrüche getrennten DNA-Ketten werden in einem letzten Schritt durch DNA-Ligase kovalent zu einem durchgehenden Tochterstrang verbunden. Der aus zahlreichen Proteinen zusammengesetzte Komplex des Primosoms bewegt sich, der Replikationsgabel folgend, unter Energieverbrauch (ATP-Hydrolyse) entlang dem template-Strang der verzögert synthetisierten DNA in 5'-3'-Richtung. In Abständen von ca. 1000 Nucleotidpositionen synthetisiert die im Primosom enthaltene Primase die kurzkettigen RNA-primer. Die Richtung dieser RNA-Synthesen ist 5'-3' bezüglich der RNA-Produkte, jedoch 3'-5' bezüglich der template-DNA. Die Bewegung des Primosoms ist daher während der kurzen Synthesephasen zumindest für die Primase-Komponente rückläufig. Die durch Helicase katalysierte Entspiralisierung der beiden DNA-Stränge an der Replikationsgabel führt zu einer Zunahme der superhelikalen Verdrillung im noch nicht replizierten (oberen) Teil der DNA. Dieser sog. superhelikale Twist würde die weitere Entspiralisierung und damit die Replikation blockieren. Er wird jedoch durch die Wirkung von Gyrase (DNA-Topoisomerasen) laufend entspannt.
Replikation
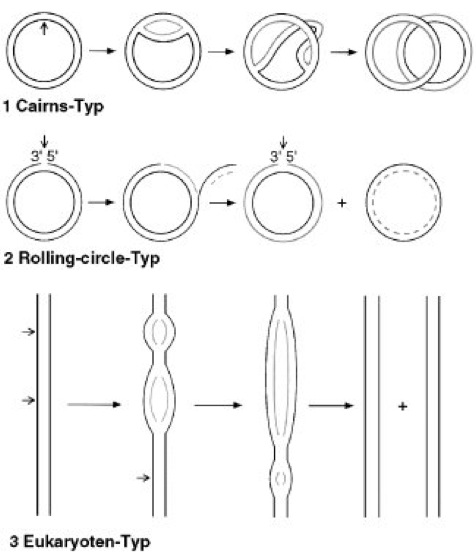
Abb. 3:Replikationstypen
Aufgrund unterschiedlicher Strukturen, die während der DNA-Replikation verschiedener Organismen beobachtet werden, unterscheidet man im allgemeinen folgende Replikationstypen (kurze Pfeile zeigen Replikationsstartpunkte an). 1Cairns-Typ oder Theta-Typ: innerhalb doppelsträngiger ringförmig geschlossener DNA bewegen sich, von einem Replikationsursprung ausgehend, 2 Y-förmige Replikationsgabeln voneinander weg (Beispiel: E. coli). 2Rolling-circle-Typ oder Sigma-Typ: ein einzelsträngiges ringförmig geschlossenes DNA-Genom wird zunächst in ein doppelsträngiges Molekül umgewandelt. Von dieser replikativen Form (RF-Form) ausgehend wird nach dem rolling-circle-Prinzip das Genom repliziert (Beispiel: einzelsträngige DNA-Phagen, z.B. ΦX174). 3Eukaryoten-Typ: von zahlreichen Replikationsstartpunkten ausgehend, wandern je 2 Replikationsgabeln in entgegengesetzter Richtung auseinander.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.