Lexikon der Biochemie: Aptamere
Aptamere, von lat. aptus, hochaffine RNA- und DNA-Oligonucleotide, die aufgrund ihrer spezifischen räumlichen Struktur eine hohe Affinität zu einem Zielmolekül besitzen. Um solche A. zu finden, wird seit Anfang der neunziger Jahre ein kombinatorischer Ansatz genutzt, bei dem nach dem Zufallsprinzip eine große Zahl von Oligonucleotiden unterschiedlichster Sequenz und Sekundärstruktur enzymatisch erzeugt wird, aus der das Oligonucleotid mit der höchsten Affinität zu einem Zielmolekül herausgesucht und angereichert wird. Als kombinatorischer Ansatz hat sich die in-vitro-Selektion bewährt. Zielstrukturen für A. können Rezeptoren und andere Proteine, Lipid- oder Kohlenhydratstrukturen sein. Interessanterweise wurden auch schon hochaffine A. gegen kleine Moleküle erhalten (Abb.). Im Gegensatz zur zeitlich und ökonomisch sehr aufwendigen konventionellen Entwicklung niedermolekularer Wirkstoffe, wie z.B. von synthetischen Agonisten für bestimmte Rezeptoren, lassen sich A. in wenigen Wochen bis Monaten erhalten. Außerdem besitzen A. selbst im Vergleich zu monoklonalen Antikörpern in der Regel viel höhere Affinitäten. Eingeschränkt werden die Verwendungsmöglichkeiten von A. allerdings durch zwei wichtige Punkte: 1) Ihre Größe wirkt limitierend für einen intrazellulären Einsatz. Zur Erzeugung der optimalen Vielfalt dreidimensionaler Strukturen von DNA- bzw. RNA-Oligonucleotid-Bibliotheken ist eine Zufallssequenz von mindestens 25 Nucleotiden erforderlich. Da für die Stabilisierung der Sekundärstruktur noch zusätzliche terminale Primer-Sequenzen nötig sind, werden die A. so groß, dass sie nur schwer in Zellen aufgenommen werden können. Aus diesem Grunde dominiert die Generierung von A., die gegen extrazelluläre Zielmoleküle gerichtet sind. 2) Ein weiterer Nachteil ist, dass bisher vorrangig RNA-Oligonucleotid-A. entwickelt wurden, da diese im Vergleich zu DNA-A. Raumstrukturen höherer Komplexität ausbilden. Andererseits ist aber RNA gegen einen Abbau durch Nucleasen weniger stabil als DNA. Durch den Einbau von 2'-modifizierten Ribosebausteinen (Ersatz des OH gegen NH2 bzw. F) wird versucht, die Abbauresistenz zu erhöhen. Allerdings müssen derartige Modifikationen auch von den Polymerasen bei der in-vitro-Selektion toleriert werden. Da auch einzelsträngige DNA zur Ausbildung dreidimensionaler Strukturen befähigt ist, wird der Entwicklung von DNA-A. große Aufmerksamkeit geschenkt. [A.D. Ellington Current Biology 4 (1994) 427; L. Gold J. Biol. Chem. 270 (1995) 13.581; L. Gold et al. Annu. Rev. Biochem. 64 (1995) 763]
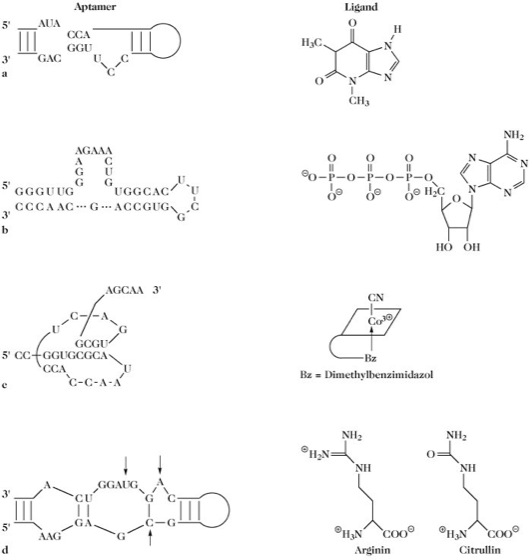
Aptamere. Beispiele einiger Aptamere.
a) Das Theophyllin-Aptamer und die Struktur von Theophyllin. Linien im Aptamer verdeutlichen, wo dessen Sekundärstruktur, aber nicht die Sequenz entscheidend für die Bindung ist (Dissoziationskonstante KD = 0,32μM).
b) Das ATP-Aptamer und dessen Ligand (KD = 0,7μM).
c) Das Cyanocobalamin-Aptamer und dessen Ligand. Linien verdeutlichen die intramolekularen Wechselwirkungen in der komplizierten Pseudoknotenstruktur (KD = 0,088μM).
d) Das Arginin-Aptamer, das aus einem Citrullin-Aptamer evolviert wurde, und beide Liganden. Pfeile deuten auf die Punkte, die für die Bindung an Arginin bzw. Citrullin bestimmend sind (KD = 10,0 μM).
Schreiben Sie uns!