Lexikon der Biochemie: DNA-Fingerprinting
DNA-Fingerprinting, DNA-Profiling, eine Methode, die auf dem Restriktionsfragment-Längenpolymorphismus (RFLP) basiert. Danach besitzt jedes Individium ein charakteristisches Restriktionsmuster in Form kurzer, meist 2-3bp langer, hoch repititiver Sequenzen (im folgenden als Monomer bezeichnet), deren Wiederholungszahlen sich stark unterscheiden (etwa 4-40). Die Detektion solch hoch variabler RFLPs ist für die Identifizierung von Individuen, wie z.B. für Täter- bzw. Vaterschaftsnachweise sehr nützlich.
Diese RFLP-Sequenzbereiche werden als Minisatelliten-DNA, Tandemwiederholungen variabler Anzahl (engl. variable number tandem repeats, VNTRs), hypervariable Loci und hochvariable Sequenzwiederholungen (engl. highly variable repeats, HVRs) bezeichnet. Die Bezeichnung "variabel" in diesen Namen weist auf die Tatsache hin, dass die Anzahl der tandemartig wiederholten Monomere an einem bestimmten genetischen Ort bei verschiedenen Individuen dieser Spezies unterschiedlich ist. Diese Anzahl kann sogar bei demselben Individuum in einem Paar homologer Chromosomen unterschiedlich sein. Die Monomere selbst scheinen aufgrund ihrer Länge und Nucleotidsequenz in verschiedene Klassen zu fallen. An einem gegebenen Ort haben die Monomere jedoch typischerweise die gleiche Nucleotidsequenz. Allerdings sind auch Abweichungen von diesem Muster bekannt. Bei Menschen sind die verschiedenen Monomerklassen dadurch charakterisiert, dass sie G-reich sind und mehrere Sequenzen besitzen, die sich ähnlich genug sind, dass eine Gensonde (Sonde) konstruiert werden kann, die unter Bedingungen niederer Stringenz an alle bindet.
Die funktionelle Bedeutung dieser hypervariablen Genorte ist noch nicht bekannt. Sie haben sich für die Identifizierung eines bestimmten Individuums, für die Bestimmung familiärer Verwandtschaft und für ökologische sowie evolutionäre Untersuchungen als sehr wertvoll erwiesen. Dies beruht darauf, dass 1) die Variation in der Monomerenanzahl an einem gegebenen hypervalenten Locus im Genom bei einer gegebenen Art übereinstimmt, 2) die hohe Zahl an hypervariablen Loci im Genom einer gegebenen Spezies übereinstimmt und 3) die hypervariablen Loci auf die gleiche Weise vererbt werden wie Mendelsche Gene.
In welcher Weise sich Individuen in ihren hypervalenten Genorten unterscheiden, wird mit Hilfe der Restriktionsanalyse chromosomaler DNA untersucht. Dies ist möglich, weil die Restriktionsstellen vieler Restriktionsendonucleasen nicht in der Minisatelliten-DNA vorkommen. Das bedeutet, dass bei einer Verdauung der chromosomalen DNA mit einer solchen Endonuclease alle Arten an Minisatelliten-DNA intakt bleiben und die Spaltungen an ihren jeweiligen Enden stattfinden. Auf diese Weise erhält man von Individuen, die an spezifischen hypervariablen Genorten tandemartige Monomerwiederholungen in unterschiedlicher Anzahl besitzen, unterschiedlich lange Restriktionsfragmente dieses Ortes. Diese können mit Hilfe der Gelelektrophorese getrennt und im Southern-Blot mit einer passenden Sonde nachgewiesen werden.
Die Vorgehensweise ist wie folgt: Die DNA-Quelle, die ausreichen sollte, um mindestens 60 ng zu isolieren, wird über Nacht mit einer Mischung aus Natriumdodecylsulfat (welches den Zellkern lysiert), Proteinase K (das die nachfolgende DNA-Gewinnung durch Verdauung des anwesenden Proteins unterstützt) und Dithiothreitol (das die Wirkung der Proteinase K durch Reduktion der Disulfidbindungen der Proteine unterstützt) inkubiert. Für forensische oder diagnostische Zwecke kann die DNA-Quelle aus Blut oder einem Blutfleck (d.h. den Leucocyten), ganzen Spermien oder einem Spermienfleck (d.h. Spermienzellen), Haarwurzelzellen, Epithelzellen aus der Mundhöhle oder Zellen aus irgendeinem verfügbaren Körpergewebe bestehen. Die DNA wird extrahiert und dann mit einer Endonuclease inkubiert, die sie in Fragmente spaltet. Von den vielen Endonucleasen, die für diesen Zweck geeignet wären, werden gewöhnlich diejenigen ausgewählt, die eine Sequenz aus vier bp erkennen (z.B. HaeIII, die 5'GG↓CC3' erkennt). Der Grund dafür ist, dass bei einer zufälligen Verteilung der Basen in der DNA eine bestimmte 4-Basen-Sequenz (z.B. GGCC) im Durchschnitt alle 44 = 256 Basenpaare vorkommt, was als eine hinreichende Spaltungsfrequenz betrachtet wird. Die DNA-Fragmente, die die hypervariablen Genorte enthalten, werden dann an Hand ihrer Größe durch Agarose-Gelelektrophorese getrennt und auf Nitrocellulosepapier übertragen (Southern-Blot). Dadurch entsteht das gleiche Bandenmuster wie es auf dem Gel vorhanden war. Das Nitrocellulosepapier wird dann in eine Lösung getaucht, die eine markierte Sonde für den gesuchten hypervariablen Genort bzw. die gesuchten Genorte enthält. Die Sondenmoleküle binden nur an solche Banden, die aus denaturierten DNA-Fragmenten zusammengesetzt sind, die ein oder mehr Monomere des hypervariablen Ziellocus bzw. der -loci enthält. Der Nachweis der Banden erfolgt mit Hilfe der Markierung, die in der Sonde vorhanden ist (z.B. Autoradiographie bei einer radioaktiv markierten Sonde). Die heute üblichen Einzel-Locussonden markieren nur eine spezifische Art eines hypervariablen Locus. Sie detektieren auf dem Southern-Blot weit weniger Banden als eine Multi-Genortsonde. Dies wird gewöhnlich dadurch kompensiert, dass die Restriktionsfragmentprobe mit mehreren verschiedenen Einzel-Locussonden separat untersucht wird.
Die diagnostischen Möglichkeiten des DNA-Fingerprinting, die erstmals 1985 aufgezeigt wurden [A.J. Jeffreys et al. Nature314 (1985), 67-73], werden in der Abb. dargestellt. Diese geben einen spezifischen hypervariablen Genort wieder, der durch ein Paar Restriktionsschnittstellen begrenzt ist (durch die Kastenform veranschaulicht) und in einem homologen Chromosomenpaar bei vier menschlichen Individuen vorkommt: A und B sind Vater und Mutter von C und D, die keine eineiigen Zwillinge sind. A und B sind beide heterozygot bezüglich des Genorts, A besitzt vier Tandemwiederholungen des Monomeren (dargestellt durch die Pfeile) auf einem Homologen (a1) und sieben auf dem anderen (a2), während B zwei (b1) und fünf (b2) besitzt. Die Kinder C (mit vier und zwei Wiederholungen) und D (mit 7 und 5 Wiederholungen) repräsentieren zwei (a1 und b1; a2 und b2) der vier möglichen genetischen Kombinationen dieses Genorts in den Nachkommen von A und B. Die beiden anderen möglichen Kombinationen sind a1 und b2 sowie a2 und b1. Der rechte Teil der Abb. zeigt die Banden des Southern-Blots, die von diesem Genort im Genom jedes dieser vier Individuen herrühren und demonstriert klar, dass das Verfahren zwischen diesen unterschieden hat. Jedoch genügt die Untersuchung eines einzigen hypervariablen Locus nicht, um C und D mit Sicherheit voneinander unterscheiden zu können. Es gibt eine Chance von 1 zu 16, dass C und D die gleiche genetische Ausstattung an dem in Frage kommenden hypervariablen Locus aufweisen. Diese Chance wird jedoch exponenziell kleiner, wenn die Anzahl der hypervariablen Loci linear erhöht wird. Die Sonden vom multi-Typ oder ein Satz aus einfach-Typen detektieren oft zwanzig oder mehr hypervariable Loci. Das Resultat stimmt mit der früheren Behauptung überein, dass A und B die Eltern der beiden Kinder sind, weil C und D die eine oder andere der beiden Banden sowohl von A als auch von B haben. Dies reicht jedoch nicht aus, um eine Elternschaft zu beweisen. Wenn jedoch das Kriterium an zwanzig oder mehr verschiedenen hypervariablen Loci erfüllt ist, wird die Möglichkeit, dass A und B nicht die Eltern von C und D sind, verschwindend gering. Die Spalte E in Abb. 1 zeigt die Banden des Southern-Blots, die sich vom hypervariablen Genort im Genom eines fünften Individuums E ableiten, das das in einer früheren Ehe geborene Kind von B ist. Die Tatsache, dass keines der Fragmentcharakteristika dieses Genorts, nämlich a1 mit vier Tandemwiederholungen und a2 mit sieben, in der Spalte E vorhanden ist, schließt A als Vater von E aus. Dies demonstriert den Wert des DNA-Fingerprintings bei Vaterschaftsverfahren.
Die Anwendbarkeit des DNA-Fingerprintings in der Forensik wurde von Gill begründet [P. Gill et al. Nature318 (1985) 577-579; P. Gill et al. Electrophoresis8 (1987) 38-44]. Das Verfahren wurde 1986 erstmals in einem Kriminalfall eingesetzt [P. Gill u. D.J. Werrett For. Sci. Int. 35 (1987) 145-148]. Es führte nicht nur zu der Schuldigsprechung eines Mannes wegen Vergewaltigung und Mord an zwei Mädchen (1983 und 1986 nahe Leicester, GB), sondern auch zum Ausschluss eines Mannes aus beiden Untersuchungen, der den Mord von 1986 gestanden hatte und von dem die Polizei deshalb glaubte, dass er auch den Mord 1983 begangen habe.
Jeffreys führt eine Fragmentübereinstimmungsstatistik von 0,25 an, d.h. die Möglichkeit, dass zwei nicht verwandte Individuen (z.B. ein Vergewaltiger und ein unschuldiges Individuum) bei zehn im D. übereinstimmenden Fragmenten das gleiche Fragmentmuster besitzen, ist 0,2510 = 9,53·10-7 oder ca. eins zu einer Million [Biochem. Soc. Trans.15 (1987) 309-317]. Obwohl dies kein absoluter Beweis ist, reicht das Verfahren möglicherweise für das Erzielen eines Schuldspruchs aus, wenn es zu allen anderen Indizienbeweisen hinzugefügt wird. Es ist offensichtlich, dass das Gewicht des DNA-Fingerprints beträchtlich steigt, wenn sich die Anzahl der gemeinsamen Fragmente erhöht.
Das Problem einiger forensischer Proben liegt darin, dass zu wenig DNA (<60 ng) zur Verfügung steht und dass sie durch Alterung und/oder andere Bedingungen, denen sie ausgesetzt war, in kurze Stücke abgebaut wurde. Es kann größtenteils durch Einsatz der Polymerasekettenreaktion (PCR) gelöst werden, mit deren Hilfe vorselektierte bekannte hypervariable Loci vervielfacht werden, die anschließend der DNA-Fingerprintanalyse unterworfen und mit den äquivalenten Loci des Verdächtigen verglichen werden. Es muss sorgfältig darauf geachtet werden, dass eine Kontamination der DNA mit DNA von Polizisten, Laboranten oder anderen Personen vermieden wird. Um diese Möglichkeit auszuschließen, wurden strenge Richtlinien aufgestellt. Beispielsweise wird die Beweis-DNA in einem anderen Labor analysiert als die DNA von einem Verdächtigen.
Vor einiger Zeit wurde ein weiteres DNA-Fingerprinting-Verfahren beschrieben [A.J. Jeffreys et al. Nature354 (1991) 204-209], das auf der Sequenzvariation in hypervariablen Loci basiert und nicht auf der Anzahl der vorhandenen Tandemwiederholungen. Es wurde erfolgreich auf den hypervariablen Genort D1S8 angewandt und überwindet viele Beschränkungen des konventionellen DNA-Fingerprintings.
Das DNA-Fingerprinting wird auf dem Gebiet der Populationsgenetik nichtmenschlicher Spezies immer häufiger eingesetzt. Die häufigsten Anwendungen sind: 1) Aufklärung des individuellen männlichen Reproduktionserfolgs in einem gegebenen Gebiet bei Wild-, Säugetier-, Vogel- und Reptilienarten, in der sich die Weibchen während ihrer fruchtbaren Phase mit mehreren verschiedenen Männchen paaren können oder fähig sind, für eine lange Zeit lebensfähige Spermien zu speichern, z.B. bei der Bestimmung, ob es eine Beziehung zwischen der Anzahl der Nachkommen, die vom Revierhalter gezeugt wurden, und der Größe seines Reviers gibt [H.L. Gibbs Science250 (1990) 1.394-1.397]. 2) Bestimmung genetischer Verwandtschaft insbesondere in der Erhaltungsgenetik [B. Amos u.a.R. Hoelzel Biol. Conserv. 61 (1992) 133-144] durch Messung des "Bandenübereinstimmungskoeffizienten" [M. Lynch Med. Biol. Evol. 7 (1990) 478-484]. Letzterer ist ein Index für die Ähnlichkeit zweier DNA-Fingerprint-Muster und wird durch die Gleichung Sxy = 2nxy/(nx + ny) berechnet, wobei nx und ny die Zahl der Banden in den DNA-Fingerprints der Individuen x und y sind und nxy die Anzahl der Banden ist, die die beiden Muster gemeinsam haben. Je höher der Wert ist, desto größer ist die Ähnlichkeit der beiden Muster (und deshalb die genetische Verwandtschaft der beiden Individuen). Ein Wert von eins zeigt eine absolute Gleichheit an.
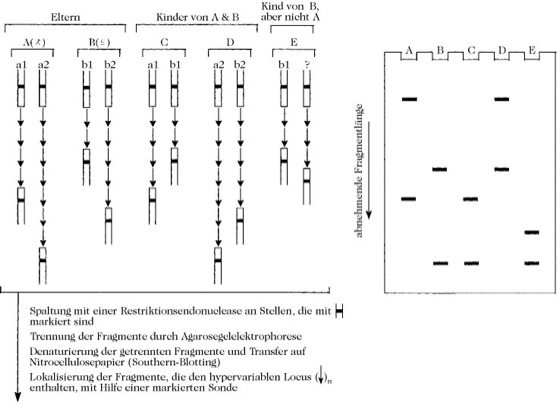
DNA-Fingerprinting. Verwendung des DNA-Fingerprintings zur Festsetzung familiärer gegenseitiger Verwandtschaft. Dieses Beispiel zeigt den DNA-Fingerprint eines hypervariablen Genorts.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.