Lexikon der Biochemie: Nucleinsäuren
Nucleinsäuren, aus Nucleotiden aufgebaute Biopolymere, die zu den wichtigsten Bestandteilen aller lebenden Zellen, Viren und Bakteriophagen gehören. N. wurden erstmals 1869 von Miescher aus Eiterzellen isoliert und als "Nuclein" bezeichnet. Den Begriff N. prägte Altmann 1889 aufgrund ihrer sauren Eigenschaften. Die zwei Hauptklassen der N. ergeben sich vor allem aus der unterschiedlichen Kohlenhydratkomponente: Ribonucleinsäure (RNA) enthält Ribose und Desoxyribonucleinsäure (DNA) 2-Desoxy-D-ribose. Beide Typen haben wesentliche strukturelle Gemeinsamkeiten, unterscheiden sich aber in ihrer Funktion. DNA dient der Speicherung der genetischen Information, die durch identische Replikation der DNA-Moleküle im Zuge der Zellteilung an die Tochterzellen weitergegeben wird. RNA vermittelt die Übertragung und Realisierung der genetischen Information in der Zelle bei der Synthese spezifischer Proteine (Proteinbiosynthese) und ist auch struktureller Bestandteil etwa der Ribosomen. Allerdings kann auch die RNA alleine Träger der genetischen Information sein, wie bei einer ganzen Reihe von Viren.
Struktur(Abb.). N. sind Polynucleotide mit Mr zwischen 20 kDa und ungefähr 106kDa. Sie bestehen aus drei verschiedenen Bestandteilen, die in charakteristischer Weise miteinander verknüpft sind: 1) die Purin- und Pyrimidinbasen Adenin, Guanin, Cytosin und Uracil (in RNA) bzw. Thymin (in DNA); darüber hinaus sind bisher in kleineren Mengen etwa 30 andere – vor allem methylierte Basen – in den verschiedensten N. nachgewiesen worden; diese seltenen Nucleinsäurebausteine entstehen durch nachträgliche Modifizierung, z.B. durch Methylierung, Hydrierung, Umlagerung der normalen Basen innerhalb der N.; 2) Monosaccharide mit fünf Kohlenstoffatomen, und zwar D-Ribose (in RNA), 2-Desoxy-D-ribose (in DNA) oder Glucose in einigen Bakteriophagen; 3) Phosphorsäure.
Die Mononucleotide, die jeweils aus einer Base, einem Zucker und einem Phosphatrest bestehen, werden durch Phosphodiesterbindungen miteinander verknüpft, wobei eine Esterbindung zwischen der 3'-Position eines Zuckers und einem Phosphatrest und die andere zwischen der 5'-Position des benachbarten Zuckers und dem Phosphatrest liegt. Diese 3',5'-Bindung führt zu einer linearen Kette in der Primärstruktur. Der Zucker ist an seinem C1 N-glycosidisch mit einer der Basen verbunden. Die lineare Anordnung der Basen ist statistisch unregelmäßig und speichert in Form des genetischen Codes die genetische Infomation des Organismus.
Die reaktiven NH2-, OH- und NH-Gruppen an Purinbasen (bzw. Pyrimidinbasen) sind für bestimmte Eigenschaften der N. verantwortlich, z.B. für die Ausbildung spezifischer Wasserstoffbrückenbindungen zwischen Purinen und Pyrimidinen, was zur Ausbildung einer räumlichen Sekundärstruktur der N. führt. Dabei können sich komplementäre lineare Ketten zu einer Doppelhelix aufrollen (DNA), oder aber ein linearer Strang faltet sich so zusammen, dass helixförmige Abschnitte mit linearen abwechseln (RNA). An der Raumstruktur der Nucleinsäuren sind aber auch homöopolare Kohäsionsbindungen (Van-der-Waals-Kräfte), hydrophobe Wechselwirkungen zwischen Basen und Lösungsmittel und elektrostatische Wechselwirkungen (lonenbindungen) beteiligt (nichtkovalente Bindungen).
Physikalische Eigenschaften und Nachweismethoden. Die konjugierten Doppelbindungen im N-heterozyklischen Ringsystem der Basen bedingen spezifische Absorptionen im UV-Licht mit einem Absorptionsmaximum bei 260 nm. UV-Spektralanalysen dienen daher zur Identifizierung und quantitativen Bestimmung der N. und ihrer Bausteine. Die Höhe der UV-Absorption ist jedoch auch von der Konformation, d.h. der Sekundärstruktur der N., abhängig. Deshalb können optische Methoden auch zur Aufklärung der Struktur und zum Nachweis von Strukturveränderungen der N. beitragen.
Bei hoher Temperatur oder bei drastischer Veränderung der Ionenkonzentration wird die Sekundärstruktur der N. zerstört (Denaturierung). Die DNA geht dabei von der Doppelhelixstruktur in den einsträngigen, geknäuelten Konformationszustand über (Helix-Knäuel-Übergang). Die Denaturierung einer N. ist von einer Zunahme der optischen Dichte (Schmelzkurve) der Lösung begleitet (hypochromer Effekt). Aus dem Verlauf der Schmelzkurve kann sowohl auf den Helixgehalt einer N. als auch auf den GC-Gehalt einer DNA geschlossen werden (Cot). Beim langsamen Abkühlen einer hitzedenaturierten N. kann es zu einer weitgehenden Wiederherstellung der ursprünglichen Struktur kommen (Renaturierung).
Zur Strukturuntersuchung der N. werden noch weitere chemische bzw. physikalische Methoden herangezogen: Hybridisierung, Elektronenmikroskopie, Ultrazentrifugationstechnik (in der analytischen Ultrazentrifuge oder in CsCl-Dichtegradienten), Röntgenstrukturanalyse, Infrarotspektroskopie, optische Rotationsdispersion, Streulichtphotometrie, Viskositätsmessungen. Die Primärstruktur von N. kann durch Nucleinsäuresequenzierung bestimmt werden.
Die Gewinnung spezifischer N. erfolgt durch Säulenchromatographie (z.B. Methylalbumin-Kieselgur-Säule), durch Elektrophorese (z.B. in Agarosegelen) und durch Dichtegradientenzentrifugation (z.B. in Saccharose oder CsCl). Zur quantitativen Bestimmung werden – neben der UV-Absorption der Basen – der Phosphatgehalt oder spezifische Farbreaktionen auf Ribose oder Desoxyribose (z.B. Feulgen-Reaktion zum histochemischen Nachweis von DNA) herangezogen.
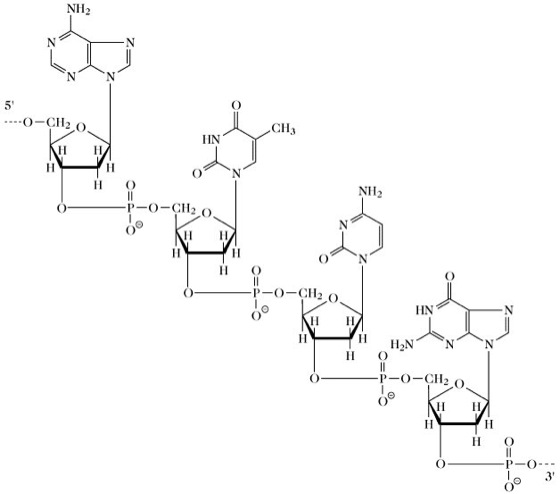
Nucleinsäuren. Polynucleotidstruktur.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.