Lexikon der Biochemie: Polymerasekettenreaktion
Polymerasekettenreaktion, PCR (engl. polymerase chain reaction), ein Prozess, der 1984 von Wissenschaftlern der Cetus Corporation entwickelt wurde und der dazu eingesetzt wird, ein spezifisches DNA-Fragment zu amplifizieren, das zwischen zwei Regionen bekannter Nucleotidsequenz innerhalb einer längeren DNA-Matrize liegt. Das Startmaterial kann sowohl aus einem einzigen DNA-Molekül als auch aus einem DNA-Molekül in einer komplizierten Mischung anderer DNA-Arten bestehen, z.B. in einem Rohzelllysat. Die Ziel-DNA ist gewöhnlich 200-500 Nucleotide lang. Zur Zeit liegt die praktische obere Grenze bei 2.000 Nucleotiden. Zusätzlich zur Matrizen-DNA werden für den Prozess zwei Oligonucleotidprimer und eine DNA-Polymerase benötigt. Die Primer sind komplementär zu den bekannten flankierenden Sequenzen der Ziel-DNA, 20-25 Nucleotide lang und werden synthetisch hergestellt. Wenn die Matrize aus einer Einzelstrang-DNA (Abb.) besteht, ist die Nucleotidsequenz eines Primers komplementär zu der bekannten Sequenz am 3'-Ende der Matrizen-DNA, während die Sequenz des anderen Primers komplementär zur äquivalenten Sequenz im komplementären Matrizenstrang ist. Wenn eine doppelsträngige Matrize vorliegt, sind die Nucleotidsequenzen der Primer komplementär zu bekannten Sequenzen, die auf den entgegengesetzten Strängen liegen, jeweils am 3'-Ende auf der Ziel-DNA.
Zu Anfang wurde in der PCR das Klenow-Fragment der E.-coli-DNA-Polymerase I als DNA-Polymerase eingesetzt. Dieses wurde später ersetzt durch die Taq-DNA-Polymerase [R.K. Saiki et al. Science239 (1988) 487-493] aus dem thermophilen Bakterium Thermus aquaticus, die ausgedehnte Inkubationen bei 95°C aushält und dadurch eine Automatisierung des Prozesses erlaubt.
Die PCR wird in mehreren Zyklen durchgeführt, wobei in jedem Durchgang die ursprüngliche Menge der in der Inkubationsmischung vorhandenen Ziel-DNA verdoppelt wird. Die Inkubationsmischung enthält außerdem einen großen molaren Überschuss der beiden Primer und der vier 2'-Desoxyribonucleotid-5'-phosphate dATP, dGTP, dCTP und dTTP. Nach dem Start der PCR werden keine Reagenzien mehr zugegeben. Der erste Schritt jedes Zyklus besteht in einer Denaturierung, wodurch die Matrizen-DNA in einsträngiger Form erhalten wird. Zur Denaturierung wird die Temperatur der Inkubationsmischung für ungefähr 60 Sekunden auf 92-96°C erhöht. Der zweite Schritt ist der Hybridisierungsschritt, bei dem die Primer an ihre komplementären Sequenzen in den einzelsträngigen DNA-Matrizenmolekülen anhybridisieren. Hierzu wird die Temperatur für ungefähr 30 Sekunden auf 55-60 °C erniedrigt. Der dritte und letzte Schritt ist der Verlängerungsschritt, bei dem die DNA-Polymerase den Primer, der durch Wasserstoffbrückenbindungen an jede Matrizen-DNA gebunden ist, in der 5'→3'-Richtung verlängert, indem sukzessiv 2'-Desoxynucleotid-5'-phosphatreste an die 3'-Hydroxylgruppe am 3'-Ende addiert werden, wobei die Sequenz durch die Matrize vorgegeben ist. Um dies zu erreichen, wird die Temperatur für 1-3 Minuten auf 72°C erhöht. Die Inkubationszeit des dritten Schritts hängt von der Länge der Ziel-DNA, die kopiert wird, ab. Bei Verwendung der Taq-DNA-Polymerase, deren Verlängerungsrate bei 70 °C 50 Nucleotide je Sekunde beträgt, ist für Ziel-DNA, die aus 500 Nucleotiden oder weniger besteht, eine Minute ausreichend. Dieser Zyklus aus drei Schritten wird viele Male wiederholt. Hierdurch wird eine exponenzielle Vervielfältigung der Ziel-DNA erreicht, weil jeder Zyklus theoretisch die Matrizenanzahl verdoppelt, die dann für den folgenden Zyklus zur Verfügung steht. Betrachtet man eine Sequenz 1-2-3-4-5 mit der komplementären Sequenz 1'-2'-3'-4'-5', bei der die Bereiche 2 und 4 bekannt sind und der Bereich 3 die Ziel-DNA darstellt, so werden die Sequenzabschnitte 2' und 4 als Primer verwendet. Damit ergibt sich folgendes Amplifikationsmuster: Am Ende jedes Zyklus liegen drei Arten an DNA-Strängen vor: 1) die ursprüngliche Matrize (1-2-3-4-5 bzw. 1'-2'-3'-4'-5'), 2) solche, die einen der beiden Primer an ihrem 5'-Ende tragen und ein 3'-Ende besitzen, das bis zum 5'-Ende der ursprünglichen Matrize verlängert wurde (2-3-4-5, ausgehend von Primer 2' bzw. 1'-2'-3'-4', ausgehend von Primer 4) und 3) solche, die exakt die Länge der Ziel-DNA plus der flankierenden Primer-DNA haben, weil sie einen der beiden Primer an ihrem 5'-Ende tragen und ihr 3'-Ende nur bis zum Ende der Sequenz verlängert wurde, die komplementär zum anderen Primer ist (2-3-4 bzw. 2'-3'-4'). Am Ende der Zyklen 1, 2 und 3 ergibt sich für diese DNA-Fragmente ein Verhältnis von 1 : 1 : 0, 1 : 2 : 1 und 1 : 3 : 4. Für die Zyklen 4, 5 und 6 liegen sie im Verhältnis 1 : 4 : 11, 1 : 5 : 26 und 1 : 6 : 57 vor. Die PCR bewirkt also keine Amplifikation der ursprünglichen Matrize, eine arithmetische Zunahme von Fragmenten, die am einen Ende der ursprünglichen Matrize entsprechen (2-3-4-5 bzw. 1'-2'-3'-4'), aber eine exponenzielle Amplifikation der Ziel-DNA mit ihren flankierenden Primersequenzen. Nach 20 Zyklen liegen die Amplifikationsprodukte im Verhältnis 1 : 20 : 1.048.555 vor und bestehen zu 99,998% aus Ziel-DNA.
Die Anzahl der benötigten thermischen Zyklen (die inzwischen in automatisierten Thermocyclern durchgeführt werden können) hängt von der Konzentration der Ziel-DNA in der Inkubationsmischung ab. Zur Amplifikation einer einzigen Kopie der Ziel-DNA in der Säuger-Genom-DNA zu einer Menge, die direkt mit Hilfe von Agarose- oder Polyacrylamidgelen bestimmt werden kann, sind mindestens 25 Zyklen notwendig. Der Wirkungsgrad der Amplifikation, der mit 100 % beginnt (d.h. jedes Matrizenmolekül in der Inkubationsmischung wird während eines gegebenen thermischen Zyklus vollständig repliziert), fällt ab und bildet ein Plateau bei 30 %, wenn die Anzahl der Zyklen 30 übersteigt. Hierfür bestehen mindestens zwei Gründe: 1) wenn die Konzentration der Produkt-DNA hoch wird, besteht eine erhöhte Konkurrenz zwischen dem Anhybridisieren der Primer an ihre Matrizen (d.h. der Produkt-DNA) und der Hybridisierung der komplementären Stränge der Matrizen und 2) die Menge der DNA-Polymerase in der Inkubationsmischung reicht eventuell nicht mehr aus, um alle Primer-Matrize-Komplexe in der Zeit zu verlängern, die für den Verlängerungsschritt des Zyklus zur Verfügung steht. Der durchschnittliche Wirkungsgrad einer Reihe von PCR-Zyklen kann mit Hilfe der Gleichung N = n(1 + E)c berechnet werden, wobei N = Menge an Ziel-DNA, die durch die Zyklenreihe gebildet wurde, n = die anfängliche Menge an Ziel-DNA, E = der Amplifikationswirkungsgrad (1 = 100 %) und c = Anzahl der Zyklen [N. Arnheim u. H. Erlich Annu. Rev. Biochem. 61 (1992) 131-156].
Die hohe Spezifität, mit der die PCR nur die Ziel-DNA amplifiziert, hängt von der Konstruktion und der Synthese der beiden Primer ab. Die Länge der Primer muss so beschaffen sein, dass ihre Sequenzen innerhalb der komplexen Mischung an DNA, die als PCR-Startmaterial verwendet werden kann, einmalig sind, und die deshalb nur mit den komplementären Sequenzen am 3'-Ende der Ziel-DNA-Stränge hybridisieren. Zwei Ansätze, die zum Ziel haben, das Problem zu minimalisieren und sicherzustellen, dass nur die Ziel-DNA amplifiziert wird, sind unter der Bezeichnung "nesting" [K.B. Mullis u. F.A. Faloona Methods Enzymol. 155 (1987) 335-350] und "hemi-nesting" [H. Li et al. Proc. Natl. Acad. Sci. USA 87 (1991) 4.580-4.584] bekannt. Bei beiden Strategien werden zwei Durchläufe der PCR mit zwei Primersätzen durchgeführt. Ein kleiner aliquoter Teil des Amplifikationsprodukts aus dem ersten Durchlauf mit dem ersten Primersatz wird als Startmaterial für den zweiten Durchlauf mit dem zweiten Primersatz verwendet. Im ersten Verfahren liegen die Sequenzen, die komplementär zum ersten Paar an Primern sind, außerhalb von jenen, die komplementär zum zweiten Primerpaar sind, dessen Abstand voneinander die Größe des amplifizierten Endprodukts bestimmt. Im zweiten Verfahren werden nur drei verschiedene Primer eingesetzt, zwei als erstwirkendes Paar, das im zweiten Durchlauf der Amplifikation durch den dritten Primer ersetzt wird. Eine neuere Modifikation des hemi-nesting-Verfahrens macht die Durchführung der beiden PCR-Durchläufe in getrennten Röhrchen unnötig. Alle drei Primer sind gleichzeitig vorhanden, die thermischen Zykluszustände werden so eingestellt, dass im ersten Durchgang die Anhybridisierung des äußerlich gelegenen Paars begünstigt wird und im zweiten Durchgang der eingebettete Primer [H.A. Erlich et al. Science252 (1991) 1.643-1.651]. Diese Einbettungsstrategien machen sich zunutze, dass jede Nichtzielsequenz, die im ersten Durchlauf amplifiziert wurde, nicht mit einem Paar an innerhalb gelegenen, zielspezifischen Primern oder sogar einem einzigen solchen Primer amplifiziert werden kann.
Bei der Verwendung von Enzymen wie der Taq-DNA-Polymerase, die keine 3'→5'-Exonuclease-Aktivität besitzt, werden häufiger fehlerhafte Produkte gebildet, bei denen einzelne falsche dNTP-Reste eingebaut wurden.
Polymerasen mit einer solchen 3'→5'-proof-reading-Aktivität und für die PCR ausreichender Thermostabilität sind z.B. die Pfu-Polymerase und die Pwo-Polymerase.
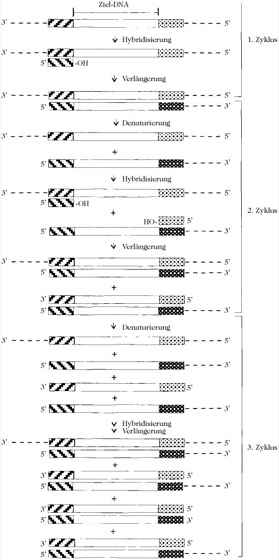
Polymerasekettenreaktion. Das Schema zeigt die ersten drei Zyklen der Amplifikation einer einsträngigen Ziel-DNA durch die Polymerasekettenreaktion. Die durch Kästchen gekennzeichneten Bereiche entsprechen komplementären DNA-Sequenzen von etwa 20 Nucleotiden Länge.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.