Lexikon der Biochemie: Replikation
Replikation, Reduplikation, der enzymatisch katalysierte Prozess der identischen und sequenzgetreuen Verdopplung von DNA. Die R. verläuft semikonservativ (Meselson-Stahl-Experiment), d.h., dass an beiden Strängen der DNA-Doppelhelix jeweils ein neuer Tochterstrang synthetisiert wird (Abb. 1).
Die R. hängt von der Zusammenarbeit von 20 oder mehr Enzymen und Proteinen ab. Die verschiedenen aufeinanderfolgenden Schritte der R. schließen die Erkennung des Ursprungs bzw. Startpunkts des Prozesses, Entwindung der Elternduplex (durch Helicasen, Topoisomerasen), Aufrechterhaltung der Strangtrennung im Replikationsbereich, Initiation der Tochterstrangsynthese, Elongation der Tochterstränge, Wiederverwindung der Doppelhelix durch Topoisomerasen und Termination des Prozesses ein. Der Komplex an Faktoren, der an der R. beteiligt ist, wird DNA-Replikase-System bzw. Replisom (Tab.) genannt.
Aufgrund von Autoradiographieuntersuchungen ist bekannt, dass die Synthese simultan vom 3'-Ende des einen Strangs und vom 5'-Ende des anderen verläuft. Jedoch arbeiten alle drei bekannten DNA-Polymerasen nur in der 5'→3'-Richtung. Der 3'→5'-Elternstrang wird in kleinen Stücken (ungefähr 2.000 Nucleotide bei Bakterien, weniger als 200 in Tierzellen) repliziert. Diese kurzen Stücke, auch Okazaki-Fragmente genannt, werden später durch eine DNA-Ligase verknüpft. Jedes Okazaki-Fragment wird als Verlängerung eines kurzen RNA-Primers (ungefähr zehn Nucleotide) synthetisiert (Abb. 2). Der RNA-Primer wird in 5'→3'-Richtung entlang der Matrize des replizierenden DNA-Strangs mit Hilfe einer spezialisierten RNA-Polymerase, der Primase, hergestellt. Die Primase ist in einem Primosomenkomplex mit anderen Proteinen assoziiert, der ebenfalls als Teil des Replisomkomplexes betrachtet werden kann (Tab.). Die DNA-Synthese geht vom 3'-Ende des Primers aus. Der RNA-Primer wird anschließend abgelöst. Dabei wird durch die 5'→3'-Exonucleaseaktivität der DNA-Polymerase I ein Nucleotid nach dem anderen abgespalten. Jedes Ribonucleotid wird nach seiner Entfernung mit Hilfe der Polymeraseaktivität desselben Enzyms durch das entsprechende Desoxyribonucleotid ersetzt. Das abschließende Zusammenfügen der Okazakifragmente wird durch die DNA-Ligase ausgeführt, die die Synthese der Phosphodiesterbindung zwischen der 3'-Phosphatgruppe der wachsenden DNA und der 5'-OH-Gruppe des neu synthetisierten Okazaki-Fragments katalysiert. Die Verknüpfung ist mit der Spaltung einer Pyrophosphatbindung gekoppelt (bei Bakterien wird NAD zu Nicotinamidmononucleotid und AMP gespalten; in Tierzellen wird ATP in AMP und Pyrophosphat überführt; Abb. 2).
Theoretisch ist die diskontinuierliche Synthese für das Fortschreiten der R. entlang des replizierenden 3'→5'-Elternstrangs nicht notwendig. Es gibt jedoch Beweise, dass dieser Vorgang ebenfalls diskontinuierlich verläuft. In einigen Fällen kann zusätzlich zu der normalen DNA-abhängigen DNA-Synthese eine RNA-abhängige Synthese erfolgen (reverse Transcriptase). [A. Kornberg DNA Replication, W.H. Freeman u. Co., 1980; A. Kornberg Supplement to DNA Replication, W.H. Freeman u. Co., 1982]
Während die DNA von Mikroorganismen als Einheit repliziert wird, ist die DNA von Eukaryonten in viele replikative Regionen (Replikon) gegliedert.
Replikation. Tab. Einige Bestandteile des Replisoms von Escherichia coli.
| |||||
| Einzelstrang-DNA- bindendes Protein | 74 | 4 | Bindung an den Einzelstrang nach Öffnung der Helix | 300 | |
| Protein i Protein n Protein n' Protein n'' dnaC dnaB Primase | 66 28 76 17 29 300 60 | 3 2 1 1 1 6 1 | Primosomenbildung Initiation der Primosomenaktivität Bildung der Primer-RNA | 50 80 70 - 100 20 50 | |
| DNA-Polymerase-III-Holoenzym, bestehend aus: α ε Θ β γ δ τ | 760 140 25 10 37 52 32 83 | 2 1 1 1 1 1 1 1 | Kettenverlängerung | 20 - - - 300 20 - – | |
| DNA-Polymerase I | 102 | 1 | Ausschneiden des Primers und Ersetzen durch DNA | 300 | |
| Ligase | 74 | 1 | Verknüpfen erweiterter Okazaki-Fragmente | 300 | |
| Gyrase, bestehend aus: Gyr A Gyr B | 400 210 190 | 4 2 2 | Superspiralisierung | - 250 25 | |
| Helicase I Helicase II | 65 75 | 1 1 | Helicase Helicase | 50 5.000 | |
| dnaA | 48 | – | Bindung an den Ursprung (origin) der Replikation |
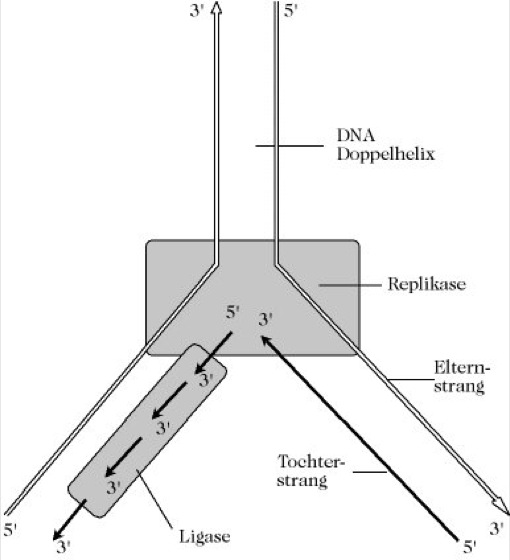
Replikation. Abb. 1. Replikationsgabel der DNA. Die Pfeile geben die Syntheserichtung an.
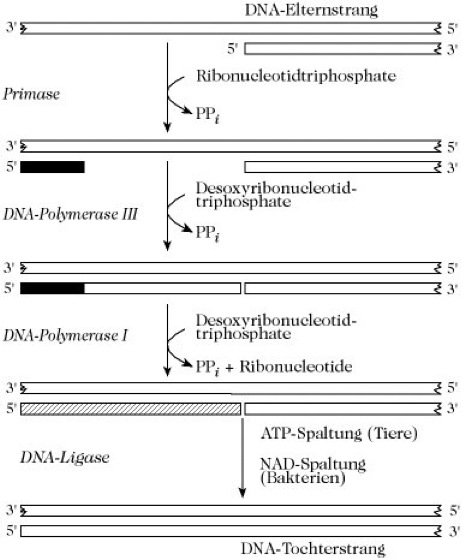
Replikation. Abb. 2. Die Rolle des RNA-Primers (schwarz) und der Okazaki-Fragmente bei der DNA-Replikation. Schwarz dargestellt ist der RNA-Primer. Der schraffiert gezeichnete Bereich entspricht einem Okazaki-Fragment.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.