Kompaktlexikon der Biologie: transfer-RNA
transfer-RNA, Abk. tRNA, kleine Ribonucleinsäure-Moleküle, die während der Translation als Adaptormoleküle („Vermittler“) zwischen der in der messenger-RNA (mRNA) gespeicherten genetischen Information und der Aminosäuresequenz des entstehenden Polypeptids fungieren. Dies ist möglich, weil für jede proteinogene Aminosäure mindestens eine, meist jedoch bis zu sechs t-RNA-Spezies existieren (so genannte Isoakzeptoren). Die hohe Spezifität wird durch die als Anticodon bezeichnete Sequenz im tRNA-Molekül ermöglicht, die alleine für den Einbau der mit der tRNA verbundenen Aminosäure verantwortlich ist (Wobble-Hypothese). Im Unterschied zu den anderen beiden RNA-Spezies kommen bei tRNAs zahlreiche Modifikationen vor, die auf Methylierung (Guanosin → 7-Methylguanosin), Desaminierung (Guanin → Inosin), Schwefelsubstitution (Uridin → 4-Thiouridin), Basenumlagerungen (Uridin → Pseudouridin) und auf die Sättigung vorhandener Doppelbindungen (Uridin → Dihydrouridin) zurückzuführen sind.
Das Vorhandensein von tRNAs wurde in den 1950er-Jahren erstmals von F. Crick vermutet; in den 1960er-Jahren gelang es R. Holley die vollständige Sequenz einer tRNA zu ermitteln.
Die Sekundärstruktur der zwischen 73 und 95 Nucleotiden umfassenden tRNA-Moleküle wird aufgrund von intramolekularen Basenpaarungen auch als Kleeblattstruktur bezeichnet und gliedert sich in bestimmte Domänen, die unterschiedliche Funktionen wahrnehmen ( vgl. Abb. ). So ist der aus den beiden Enden bestehende, sieben Basenpaare umfassende Bereich, der als Stamm oder Akzeptor-Arm bezeichnet wird, für die Anbindung der jeweiligen Aminosäure verantwortlich. Der Anticodon-Arm enthält die für die Translation erforderliche Information, wohingegen die anderen beiden Arme aufgrund des Vorhandenseins modifzierter Nucleotide als DHU-Arm (Dihydrouracil) und TΨC-Arm (Pseudouracil) bezeichnet werden. Sie dienen vor allem der Stabilisierung der tRNA-Moleküle. Die Tertiärstruktur der tRNAs wurde durch Röntgenstrukturanalyse bestimmt und wird als so genannte L-Form bezeichnet ( vgl. Abb. ).
Die Aminoacylierung oder Beladung der tRNAs erfolgt durch die Bildung einer kovalenten Bindung zwischen der Carboxylgruppe am C-Terminus der Aminosäure und einer OH-Gruppe am 3'-Ende der tRNA ( vgl. Abb. ). Diese Reaktion wird durch so genannte Aminoacyl-tRNA-Synthetasen katalysiert, wobei für jede Aminosäure ein eigenes Enzym existiert; Isoakzeptoren werden somit durch dasselbe Enzym beladen. Die Enzyme stellen eine heterogene Gruppe dar, deren Reaktion sich wie folgt beschreiben lässt:
Aminosäure + ATP + tRNA → Aminoacyl-tRNA + AMP + PPi
Dabei entsteht zunächst ein Anhydrid zwischen der Aminosäure und dem AMP; die so aktivierte Aminosäure wird anschließend auf die OH-Gruppe der Ribose am 3'-Ende der tRNA übertragen (Esterbildung). Die dabei entstehende energiereiche Bindung wird später während der Translation zur Bildung der Peptidbindung zwischen der Aminosäure und der wachsenden Polypeptidkette genutzt. Aminoacyl-tRNA-Synthetasen lassen sich in zwei Klassen einteilen, wobei Klasse I die Aminosäuren mit der 2'-OH-Gruppe der Ribose am 3'-Ende verknüpft, Enzyme der Klasse II hingegen die Veresterung mit der 3'-OH-Gruppe katalysieren.
Prokaryotische und eukaryotische tRNAs werden durch RNA-Polymerasen als Vorläufer-tRNAs (Precursor) transkribiert und anschließend einer Prozessierung unterzogen. Bei Escherichia coli kommen 80 tRNA-Gene vor, die teilweise in den Transkriptionsabschnitten für ribosomale RNA vorliegen. Für eine Reihe von tRNA's, die häufige Codons bedienen, liegt im Bakteriengenom somit mehr als eine Kopie vor.

transfer-RNA:1Sekundärstruktur („Kleeblattstruktur“) einer transfer-RNA mit den wichtigsten Merkmalen, die für die Funktion des Moleküls verantwortlich sind. 2Tertiärstruktur („L-Form“), die durch nicht eingezeichnete Wasserstoffbrücken stabilisiert wird, wodurch der Akzeptor-Arm, an den die Aminosäurebindung erfolgt, und der Anticodon-Arm an den äußersten Enden der dreidimensionalen Struktur liegen

transfer-RNA: Lineare Nucleotidsequenz eines tRNA-Moleküls. Die Lage von DHU-Arm, Anticodon-Arm, TΨC-Arm und dem CCA-Ende, an dem die Aminoacylierung erfolgt, sind von links nach rechts (5'-3'-Richtung) hervorgehoben
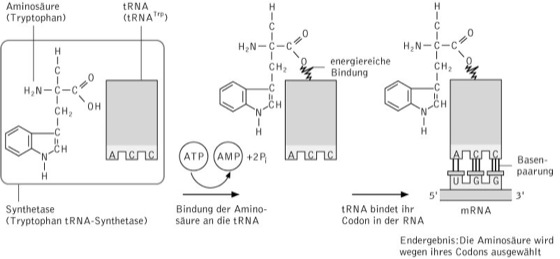
transfer-RNA: Aminoacylierung („Beladung“) einer transfer-RNA mit einer Aminosäure am Beispiel von Tryptophan. Die Spezifität der Beladung garantiert, dass während der Translation durch eine Codon-Anticodon-Erkennung die richtige, d.h. dem genetischen Code eines Gens entsprechende Aminosäure für die Proteinsynthese verwendet wird
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.