Biochemie: Erforschung der Proteine und Proteome
Eines der Hauptziele der Biochemie ist es herauszufinden, wie die Aminosäuresequenz die Konformation eines Proteins und damit seine Funktion festlegt. Wir möchten außerdem wissen, wie Proteine spezifische Substrate und andere Moleküle binden, wie sie katalytisch aktiv werden und wie sie Energie und Information übertragen.
Häufig empfiehlt sich, ein Protein erst dann zu untersuchen, wenn man es von den übrigen Zellbestandteilen abgetrennt hat, so dass Struktur und Funktion dieses Proteins ohne die störenden Effekte von Kontaminationen getestet werden können. Der erste Schritt in einer Versuchsreihe mit einem Protein besteht daher in dessen Reinigung. Proteine lassen sich auf Grund ihrer unterschiedlichen Löslichkeit, Größe, Ladung und Bindungskapazität voneinander trennen. Hat man ein Protein gereinigt, kann man seine Aminosäuresequenz bestimmen. In riesigen Sequenzdatenbanken stehen nun die Aminosäuresequenzen von zahlreichen Proteinen, die häufig auch aus Genomsequenzen abgeleitet wurden, zur Verfügung. Wenn die Sequenz eines gereinigten Proteins bereits in einer allgemein zugänglichen Datenbank enthalten ist, erleichtert das die Forschungsarbeit erheblich. Man muss nur einen kleinen Abschnitt einer Aminosäuresequenz bestimmen, um in der Datenbank das entsprechende Protein zu finden. Alternativ kann man ein Protein auch auf Grund seiner Masse identifizieren, wenn man sie mit den Massezahlen vergleicht, die aus den Aminosäuresequenzen in der Datenbank abgeleitet werden können. Die Massenspektrometrie bietet hier ein leistungsfähiges Verfahren für die Bestimmung der Masse und der Sequenz eines Proteins.
Nachdem ein Protein aufgereinigt wurde und man seine Identität kennt, ist immer noch zu klären, welche Funktion es in einem physiologisch relevanten Kontext besitzt. Am besten sind Antikörper geeignet, ein Protein in vivo zu lokalisieren und seine Menge zu bestimmen. Monoklonale Antikörper, die spezielle Aminosäuresequenzen erkennen, lassen sich in großen Mengen herstellen. Mit ihrer Hilfe kann man das Protein bei der Isolierung und in Zellen nachweisen und quantifizieren. Peptide und Proteine lassen sich chemisch synthetisieren, und man kann sie so in der Forschung und in bestimmten Fällen in hochreiner Form auch als Medikamente einsetzen. Röntgenstrukturanalysen und Kernspinresonanzspektroskopie sind die wichtigsten Methoden zur Aufklärung der dreidimensionalen Struktur und damit der für die Funktion entscheidenden Eigenschaft.
Die Erforschung von Proteinen mit den hier aufgeführten physikalischen und chemischen Methoden hat unser Wissen über die molekularen Grundlagen des Lebens beträchtlich erweitert, und sie hilft uns, einigen der faszinierendsten Fragen der Biologie auf molekularer Ebene nachzugehen.
Das Proteom ist die funktionelle Darstellung des Genoms
Wie in Kapitel 5 besprochen wird, sind heute von vielen Organismen die vollständigen Basensequenzen ihrer DNA bekannt. Der Nematode Caenorhabditis elegans verfügt über ein Genom aus 97 Millionen Basen und etwa 19 000 proteincodierenden Genen, die Taufliege Drosophila melanogaster hingegen aus über 180 Millionen Basen und etwa 14 000 Genen. Die vollständige Sequenz des menschlichen Genoms umfasst 3 Milliarden Basen mit etwa 23 000 Genen. Diese Genome sind jedoch nichts anderes als systematische Verzeichnisse von Genen, die unter spezifischen Bedingungen in einer Zelle exprimiert werden könnten. In einem bestimmten biologischen Kontext ist immer nur ein Teil der Proteine vorhanden, die von diesen Genen codiert werden. Das Proteom eines Organismus – abgeleitet aus den Proteinen, die das Genom exprimiert – bezeichnet eine komplexere Informationsebene, eine Ebene der funktionellen Information, und beinhaltet Art, Funktion und Interaktion von Proteinen innerhalb einer biologischen Umgebung.
Das Proteom ist keine festgelegte Eigenschaft der Zelle. Da es die funktionelle Expression der Information darstellt, variiert es abhängig vom Zelltyp, dem Entwicklungsstadium und den Bedingungen in der Umgebung, etwa dem Vorhandensein von Hormonen. Das Proteom ist viel größer als das Genom, da fast alle Genprodukte Proteine sind, die auf verschiedene Weise modifiziert werden können. Darüber hinaus existieren diese Proteine nicht für sich allein; sie treten häufig untereinander in Wechselwirkung und bilden so Komplexe mit spezifischen funktionellen Eigenschaften. Während das Genom festgelegt ist, erweist sich das Proteom als hochdynamisch. Verstehen lässt sich das Proteom, indem man die beteiligten Proteine untersucht, charakterisiert und auflistet. Häufig, aber nicht immer, beginnt man damit, ein spezielles Protein von allen anderen Biomolekülen der Zelle abzutrennen.
3.1 Die Reinigung eines Proteins ist der erste Schritt zum Verständnis seiner Funktion
Ein altes Sprichwort in der Biochemie rät: Verschwende nie reine Gedanken an ein unreines Protein. Mit einem reinen Protein in der Hand können wir dessen Aminosäuresequenz und biochemische Funktion bestimmen. Auf Grund der Aminosäuresequenz kann man die evolutionären Beziehungen zwischen Proteinen in verschiedenen Organismen untersuchen (Kapitel 6). Auch lassen sich von einem gereinigten Protein Kristalle und aus diesen wiederum Röntgenstrukturdaten gewinnen, die uns ein Bild von der Tertiärstruktur des Proteins vermitteln – denn die Form bestimmt die Funktion.
Der Assay: Woran erkennen wir das Protein, nach dem wir suchen?
Ziel einer Reinigung ist eine Proteinprobe, die nur noch Moleküle einer einzigen Art enthält: das Protein, das den Biochemiker interessiert. Diese Proteinprobe macht womöglich den Bruchteil von nur einem Prozent des Ausgangsmaterials aus, wobei dieses aus kultivierten Zellen oder auch aus einem speziellen pflanzlichen oder tierischen Organ bestehen kann. Wie ist ein Biochemiker in der Lage, aus einer komplexen Mischung von Proteinen ein bestimmtes Protein herauszufischen?
Ein Protein lässt sich dadurch aufreinigen, dass man das ungereinigte Gemisch des Ausgangsmaterials einer Reihe von Trennschritten unterzieht, die auf physikalischen Eigenschaften wie Größe und Ladung beruhen. Um den Erfolg dieser Reinigung messen zu können, benötigt man einen Assay, einen Test auf irgendeine besondere Eigenschaft des Proteins, der darüber Auskunft gibt, ob es in der Präparation vorhanden ist oder nicht. Einen geeigneten Assay zu entwickeln, kann eine schwierige Aufgabe sein, doch je spezifischer dieser ist, umso effizienter wird die Reinigung. Bei Enzymen, Katalysatoren in Proteingestalt (Kapitel 8), besteht der Assay in der Regel aus der Reaktion, die das Enzym in der Zelle katalysiert. Betrachten wir das Enzym Lactat-Dehydrogenase, einen wichtigen Akteur bei der anaeroben Energiegewinnung aus Glukose und bei der Synthese von Glukose aus Milchsäure. Die Lactat-Dehydrogenase katalysiert folgende Reaktion:
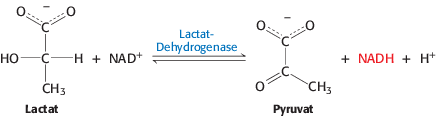
Die reduzierte Form von Nicotinamidadenindinucleotid (NADH, Abbildung 15.13, in dieser Leseprobe nicht enthalten) absorbiert Licht mit einer Wellenlänge von 340 Nanometer, was bei der oxidierten Form (NAD+) nicht der Fall ist. Demnach können wir den Fortgang der Reaktion verfolgen, indem wir beobachten, wie viel Licht das Reaktionsgemisch bei 340 Nanometer pro Zeiteinheit – beispielsweise binnen einer Minute nach Zugabe des Enzyms – absorbiert. Bei der Reinigung der Lactat-Dehydrogenase besteht unser Assay für die Enzymaktivität also in der Beobachtung des Anstiegs der Absorption beziehungsweise Extinktion bei 340 Nanometer pro Minute.
Um festzustellen, ob unser Reinigungsschema funktioniert, benötigen wir noch eine zusätzliche Information – wir müssen wissen, wie viel Protein in der getesteten Lösung vorhanden ist. Es gibt unterschiedlich schnelle und genaue Methoden zur Bestimmung der Proteinkonzentration. Mit diesen beiden experimentell bestimmten Daten – Enzymaktivität und Proteinkonzentration – können wir dann die spezifische Aktivität, also das Verhältnis der Enzymaktivität zur Proteinmenge im Enzymassay, berechnen. Mit fortschreitender Reinigung wird das Proteingemisch im Idealfall einen immer größeren Anteil des gesuchten Proteins und damit eine steigende spezifische Aktivität aufweisen. Im Prinzip besteht das Ziel der Reinigung in der Maximierung der spezifischen Aktivität. Bei einem reinen Enzym zeigt die spezifische Aktivität einen konstanten Wert.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.