Lexikon der Biochemie: kinetische Datenauswertung
kinetische Datenauswertung, eine Datenauswertung, die mit Hilfe des Computers durch eine nichtlineare Regressionsanalyse ausgeführt wird. Es wird ein objektives Verfahren angewandt, bei dem die Summen der Quadrate der Differenzen zwischen berechneten und experimentell bestimmten Werten minimiert werden (Methode der kleinsten Quadrate). Wenn eine Reaktion der Michaelis-Menten-Kinetik folgt, ergibt die graphische Darstellung der Anfangsgeschwindigkeit (v) gegen die Substratkonzentration (S) eine gleichseitige Hyperbel der Form: v = VmaxS/(Km + S). Für eine erste Näherung im Labor kann diese Gleichung so umgeformt werden, dass die Grundlage für mehrere lineare Transformationen geschaffen wird, mit deren Hilfe Vmax (Maximalgeschwindigkeit) und Km (Michaeliskonstante) bestimmt werden können. Solche graphischen Auswertungen sind nicht ganz frei von subjektiven Einflüssen.
Das Lineweaver-Burk- bzw. doppelt reziproke Diagramm (Tab. und Abb.) wird oft verwendet, es ist jedoch das am wenigsten zuverlässige der möglichen linearen Transformationen. Kleine Fehler in v für kleine Werte von v ziehen sehr große Fehler für 1/v nach sich. Für große Werte von v wird der gleiche kleine Fehler für 1/v vernachlässigbar. Die Autoren dieser Methoden wiesen auf den Fehler hin, der hohen 1/v-Werten anhaftet und auf die Notwendigkeit, die niedrigen Werte von 1/v stärker zu gewichten. Dies wird jedoch von vielen Anwendern missachtet. Es wurde vorgeschlagen, das Lineweaver-Burk-Diagramm allgemein zur Bestimmung der Km-Werte anzuwenden.
Eine zufriedenstellendere Fehlerstreuung ergibt sich bei der Auftragung von S/v gegen S, dem Hanes-Diagramm bzw. Hanes-Wilkinson-Diagramm (Tab. und Abb. C). Eine weitere lineare Transformation stellt v gegen v/S dar, das Eadie-Hofstee-Diagramm. Der Fehler nimmt mit steigendem v/S zu, da v jedoch Bestandteil beider Koordinaten ist, variiert der Fehler eher in Bezug auf den Ursprung als auf die Achsen, d.h. die Fehlerbalken laufen am Ursprung zusammen (Tab. und Abb. B).
Die zufriedenstellendste Behandlung kinetischer Daten stellt das direkte lineare Diagramm von Eisenthal und Cornish Bowden (1974) dar. Auf den Achsen werden -S als Abszisse und v als Ordinate aufgetragen. Anstatt das übliche hyperbolische Diagramm von S/v (Enzymkinetik) aufzustellen, werden zusammengehörende Punkte (jeder -S-Wert wird mit seinem v-Wert in Beziehung gesetzt) durch eine Gerade verbunden. Der Punkt, an dem sich die Geraden dieser Familie kreuzen, ergibt die Werte für Vmax und Km. Der mathematische Hintergrund dieses Diagramms ist die Umformung der allgemeinen Gleichung zu: Vmax = v + vKm/S, wobei Vmax und Km nur scheinbar Variable sind. In Wirklichkeit gehört nur ein Wert für Vmax und Km zu allen v- und S-Paaren. Aufgrund experimenteller Fehler ist der Kreuzungspunkt der Geraden nicht immer eindeutig definiert. In diesem Fall wird der Punkt gewählt, an dem die meisten Geraden zusammentreffen. Der Vorteil dieser Methode liegt darin, dass jede Messung einzeln dargestellt wird und dann als schlecht bewertet werden kann, wenn ihre Gerade nicht mit der Mehrheit übereinstimmt (Tab. und Abb. D).
Das Scatchard-Diagramm wird oft für die Bestimmung von Bindungskonstanten eingesetzt, die anderen Diagramme (A-D) sind für diesen Zweck ebenso geeignet. Solche Messungen sind wichtig, z.B. für die Bestimmung der Stärke der Assoziation zwischen Hormonen und Zellmembranen, zwischen Regulationsenzymen und Effektormolekülen und zwischen Steroidhormonen und ihren hochaffinen Rezeptorproteinen in den Zellen des Zielorgans. Das Scatchard-Diagramm stellt einen Sonderfall der Hill-Gleichung (Hill-Plot) dar, wenn nH = 1. Die Konzentration eines Bindungspartners wird konstant gehalten, wobei dies gewöhnlich ein Protein, eine Organelle oder eine Zelle sein wird. Die Konzentration des kleineren Liganden wird variiert (Tab. und Abb. E).
Kinetische Datenauswertung. Tab. Lineare Transformationen der Gleichung v = VmaxS/(Km + S), wobei v = Anfangsgeschwindigkeit, Vmax = Maximalgeschwindigkeit, wenn das Enzym mit Substrat gesättigt ist, Km = Michaelis-Konstante, S = Ausgangssubstratkonzentration. Die lineare Scatchard-Gleichung ist ebenfalls aufgeführt, mit b = Konzentration des gebundenen Liganden, f = Konzentration des freien Liganden, Kd = Dissoziationskonstante, bmax = Maximalkonzentration von b, wenn der Ligand sättigt.
| |||||||
| Lineweaver-Burk (doppelt reziprokes Diagramm; A) | |||||||
| Eadie-Hofstee (B) | V | Vmax | -Km | ||||
| Hanes-Wilkinson (C) | S | -Km | |||||
| Eisenthal-Cornish Bowden (direkt lineares Diagramm; D) | V | -S | Verbindungslinien S1 – v1, S2 – v2, Sn – vn Schnittpunkt bei Vmax und Km | ||||
| Scatchard (E) | b | bmax |
*Buchstaben in Klammern beziehen sich auf die Abb.
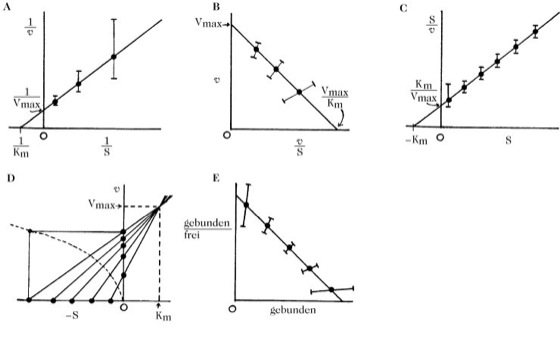
Kinetische Datenauswertung. Abb. Lineare Transformation der Gleichung v = VmaxS/(Km + S). Die Fehlerbalken sind ebenfalls eingezeichnet. A: Lineweaver-Burk-, oder doppelt reziprokes Diagramm; B: Eadie-Hofstee-Diagramm; C: Hanes-Wilkinson-Diagramm; D: Eisenthal-Cornish Bowden-, oder direkt lineares Diagramm; E: Scatchard-Diagramm, das zur Bestimmung von Bindungskonstanten herangezogen wird.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.