Lexikon der Biochemie: Maxam-Gilbert-Verfahren
Maxam-Gilbert-Verfahren, eine Methode der Nucleinsäuresequenzierung zur Sequenzbestimmung von DNA. Hierbei werden DNA-Einzelstrangfragmente, die an einem Ende, oft dem 5'-Ende, radioaktiv markiert wurden, chemisch gespalten. Zusätzlich können die DNA-Fragmente mit [χ-32P]ATP in der Gegenwart von Polynucleotid-5'-Hydroxylkinase inkubiert werden, wobei an die Hydroxylgruppe des 5'-Endes eine [32P]-Phosphatgruppe addiert wird. Wenn diese Hydroxylgruppe bereits eine Phosphatgruppe trägt, wie es bei nativer DNA der Fall ist, muss diese zuerst durch Inkubation der DNA mit alkalischer Phosphatase aus E. coli oder Kalbsdarm entfernt werden. Dann werden aliquote Teile der Präparation mit markierter DNA mindestens vier verschiedenen chemischen Verfahren unterworfen, durch die ein spezifisches Nucleosid (d.h. eine N-Base und ihre N-glycosidisch gebundene 2-Desoxy-D-ribose) oder eine spezifische Nucleotidart von der Polynucleotidkette abgespalten wird. Die Bedingungen, unter denen jedes dieser Spaltungsverfahren durchgeführt wird, werden so gewählt, dass jedes markierte DNA-Molekül in der DNA-Probe im Durchschnitt nur einmal gespalten wird, wobei die Spaltstelle zufällig lokalisiert ist. Es entstehen zwei Spaltprodukte: eines mit einem [32P]Phosphat an seinem 5'-Ende und einem nichtmarkierten Phosphat an seinem 3'-Ende und eines mit einer nichtmarkierten Phosphatgruppe an seinem 5'-Ende und entweder einer Hydroxylgruppe oder einer nichtmarkierten Phosphatgruppe an seinem 3'-Ende. Von diesen beiden Produkten wird mit Hilfe der anschließenden Autoradiographie nur das markierte detektiert. Wenn das genannte Verfahren beispielsweise auf die Sequenz (5')CGATGGCAGTCT(3') angewandt wird, entstehen bei Spaltung vor G die folgenden fünf 32P-markierten Fragmente: 32P-C, 32P-CGAT, 32P-CGATG, 32P-CGATGGCA. Dieser Satz wird dann elektrophoretisch aufgetrennt. Die analogen Fragmentsätze, die sich bei den anderen Spaltungsverfahren ergeben, befinden sich in dazu parallelen Bahnen. Auf Sequenzgelen (Elektrophorese bei ~8M Harnstoff und ~70 °C, um Wechselwirkungen über Wasserstoffbindungen zu unterdrücken) können Fragmente getrennt werden, die sich in ihrer Länge um einen Nucleotidrest unterscheiden. Die Lage der getrennten Fragmente in jeder Bahn wird durch Autoradiographie bestimmt. Aus dem Bandenmuster kann die vollständige Nucleotidsequenz der untersuchten DNA abgeleitet werden (Abb.).
Allgemein werden bei dem M. folgende vier Spaltungsreaktionen durchgeführt: 1) Spaltung bei Guanin (G). Behandlung der markierten DNA mit Dimethylsulfat in wässrigem Puffer bei pH 8 führt zur Guaninmethylierung an N-7, die C8-C9-Bindung des Guanins wird basenkatalysiert gespalten und der 2-Desoxy-D-riboserest wird durch Piperidin entfernt. 2)Spaltung bei Purinen (G und A). Die Abspaltung von G und A nutzt die Tatsache, dass unter sauren Bedingungen (pH 2) die Purin-N-Atome der markierten DNA protoniert werden, wodurch die glycosidische Bindung von G und A geschwächt wird. Dadurch wird der Weg zur Abspaltung von G und A und ihrer 2-Desoxy-D-ribosereste durch Piperidinbehandlung geebnet. 3) Spaltung bei Pyrimidinen (C und T). Zur Spaltung von C und T wird die markierte DNA mit wässrigem Hydrazin behandelt. Hierbei werden die Pyrimidinringe durch konjugierte nucleophile Addition an ihre Carbonylgruppen geöffnet. Durch Behandlung mit Piperidin werden dann C und T in Form eines fünfgliedrigen N-Heterozyklus abgespalten und ihre Ringreste anschließend als Harnstoff zusammen mit den 2-Desoxy-D-riboseresten entfernt. 4) Spaltung bei Cytosin (C). Um C allein abzuspalten, wird wie bei der Abspaltung von C und T verfahren, jedoch wird Hydrazin in Gegenwart von 1,5M NaCl eingesetzt, um eine Ringöffnung von T zu verhindern. Auch andere Spaltungsverfahren wurden beschrieben [B.J.B. Ambrose u. R.C. Pless Methods Enzymol. 152 (1987) 522-538].
Die Abb. zeigt den Satz markierter Fragmente, der aus der oben genannten DNA mit Hilfe der vier Spaltungsmethoden erzeugt wurde, nach Trennung auf einem Sequenzgel. Die nichtgespaltene, markierte DNA, die 12 Nucleotide lang ist, legt die kürzeste Wegstrecke zurück und erscheint in allen vier Bahnen an der gleichen Position als dunkle Bande. Die Position von A-Nucleotiden ergibt sich aus dem Vergleich der A + G-Spur mit der G-Spur, ebenso wie die Position der T-Nucleotide aus dem Vergleich der T + C-Spur mit der C-Spur.
Das Nucleotid am 5'-Ende der DNA kann mit Hilfe des M. nicht bestimmt werden. In der Praxis wird das zweite Nucleotid nach dem 5'-Ende (*C in der Abb.) häufig auf dem Gel auch nicht identifiziert. Die Identität dieser beiden Nucleotide kann jedoch durch Sequenzierung des komplementären DNA-Strangs festgestellt werden kann. Mit dem M. können DNA-Fragmente von bis zu 250 Nucleotiden erfolgreich sequenziert werden.
Alternativ zu dieser chemischen Methode wurde die Sanger-Sequenzierung entwickelt, die heute wesentlich häufiger angewandt wird.
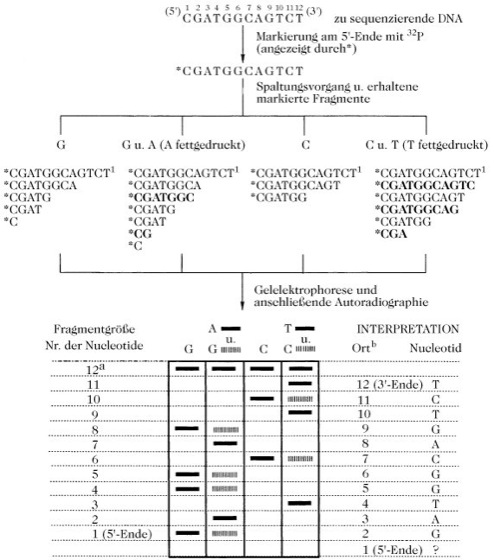
Maxam-Gilbert-Verfahren. Das Schema zeigt die Methode der chemischen DNA-Spaltung nach Maxam und Gilbert, angewandt auf die Bestimmung der Nucleotidsequenz eines hypothetischen DNA-Abschnitts. a = Ursprungs-DNA, d.h. die nichtgespaltene, jedoch 32P-markierte DNA, die sequenziert wird. b = Position jedes Nucleotids in der sequenzierten DNA, nummeriert vom 5'-Ende aus.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.