Lexikon der Biochemie: Sanger-Sequenzierung
Sanger-Sequenzierung, Didesoxymethode, Kettenabbruchmethode, eine Methode zur Sequenzierung von DNA (Nucleinsäuresequenzierung). Die S. ist heute verbreiteter als das Maxam-Gilbert-Verfahren, weil die benötigten Reagenzien jetzt gut verfügbar sind und das Verfahren automatisiert wurde.
Bei der S. katalysiert eine DNA-Polymerase die Synthese komplementärer Kopien der Einzelstrang-DNA, die sequenziert werden soll. Die Nucleotidsequenz der komplementären Kopie wird direkt bestimmt und die Sequenz der Ursprungs-DNA auf der Grundlage der Basenpaarung abgeleitet. Dies bedeutet, dass die DNA, die sequenziert werden soll, als Matrize für die Erzeugung von komplementären Kopien verwendet wird. Zu diesem Zweck wird sie in einen geeigneten Vektor (Phage oder Plasmid) inseriert und kloniert. Zur Initiation des Replikationsprozesses benötigt die DNA-Polymerase einen Primer (ein kurzes DNA-Segment), der stabile Basenpaarungen zu einer komplementären Nucleotidsequenz der Phagen- bzw. Plasmid-DNA bildet. Synthetische Primer sind ungefähr 20 Nucleotide lang und kommerziell erhältlich. Wenn die Matrize-Primer-Duplex mit den vier Desoxyribonucleosid-5'-triphosphaten (dATP, dGTP, dCTP, dTTP) in Gegenwart der geeigneten DNA-Polymerase inkubiert wird, wird der Primer fortlaufend verlängert, indem Desoxyribonucleosid-5'-monophosphatreste an die terminale 3'-Hydroxylgruppe (5'®3'-Richtung) in einer Reihenfolge addiert werden, die durch die Matrize und die Gesetze der Basenpaarung vorgegeben ist. Jede neue Phosphodiesterbindung wird durch Kondensation des α-Phosphatrests an der 5'-Hydroxylgruppe des eintretenden Desoxyribonucleotids mit der 3'-Hydroxylgruppe des terminalen Desoxyribonucleotids der sich verlängernden Kette gebildet. Die Elongation hängt deshalb vom Vorhandensein einer 3'-Hydroxylgruppe am terminalen Desoxyribonucleotid der Kette ab, die verlängert wird.
Zur Sequenzbestimmung werden vier Inkubationen durchgeführt, von denen jede die Matrize-Primer-Duplex, die passende DNA-Polymerase, gleiche Mengen der vier Desoxyribonucleosid-5'-phosphate, von denen mindestens eines im α-Phosphatrest z.B. mit 32P markiert ist, und eine kleine Menge des 2',3'-Didesoxyderivats von einem der vier Desoxyribonucleotid-5'-phosphate (ddATP, ddGTP, ddCTP oder ddTTP) enthält. Wenn in die wachsende Polynucleotidkette anstelle eines 2'-Desoxynucleotids das analoge 2',3'-Didesoxyderivat eingebaut wird, stoppt die Kettenverlängerung, weil eine 3'-Hydroxylgruppe fehlt. Die Menge des 2',3'-Didesoxynucleotids, die in jedem Inkubationsgemisch vorhanden ist, ist im Verhältnis zu jedem der 2'-Desoxyribonucleotide gering, so dass ein Satz an 32P-markierten Ketten mit unterschiedlicher Länge erzeugt wird. Wenn ddGTP eingesetzt wird und die Matrizen-DNA die Sequenz (3')TCTGACGGTAGC(5') besitzt, wird folgender Satz an 32P-markierten Ketten vom Enzym freigesetzt: Primer-(5')AddG(3'), Primer-(5')AGACTddG(3') und Primer-(5')AGACTGCCATCddG(3'). Dieser und die drei anderen Sätze an Nucleotidketten, die unter Verwendung der anderen 2',3'-Didesoxyribonucleotide erzeugt werden, werden in parallelen Bahnen eines Polyacrylamid-Sequenzierungsgels aufgetrennt. Das Bandenmuster kann dann z.B. durch Autoradiographie bestimmt werden. Die Abb. zeigt die Anwendung dieses Verfahrens auf die oben erwähnte Matrizen-DNA (3')TCTGACGGTAGC(5').
Die grundlegende Methode wurde auf verschiedene Weise variiert und verbessert, das prinzipielle Konzept blieb jedoch gleich. Die Verbesserungen betrafen folgende fünf Bereiche: 1) die Anwendbarkeit und bequeme Handhabung der Vektoren, in die die DNA, die sequenziert werden soll, eingebaut wird, 2) die Art der Markierung der 2',3'-didesoxyribonucleotidterminierten Ketten, 3) die Verwendung von 2'-Desoxyribonucleosid-5'-triphosphatanaloga, um die sog. "Komprimierung" der Banden auf dem Sequenzierungsgel zu eliminieren, 4) die verwendeten DNA-Polymerasen und 5) die Möglichkeit, sowohl doppelsträngige als auch einzelsträngige DNA-Matrizen zu sequenzieren.
Spezifische Vektoren, die so entwickelt wurden, sind Phagen (z.B. M13 und seine Konstrukte wie M13mp19), Plasmide (z.B. die pUC-Serien) und Phagemide (z.B. pBluescript→ II KS +/-) mit hoher Kopienzahl, Resistenz gegen spezifische Antibiotika, multiplen Restriktionsendonucleasespaltungsstellen und bekannten codierenden Sequenzen, in die die DNA, die sequenziert werden soll, genau eingefügt werden kann.
Zur Markierung der DNA wurde zunächst [35S]dATPαS anstelle von [32P]dATP eingesetzt. Bei dieser Verbindung bildet ein 35S-Atom eine Doppelbindung zum P-Atom des α-Phosphatrests im 2'-Desoxyribonucleotid anstelle des O – wie beim dATP – aus. Wenn mit Hilfe der DNA-Polymerase ein dAMP-Rest in die wachsende DNA-Kette eingebaut wird, wird diese aufgrund der Bildung einer atypischen Phosphodiesterbindung [R-O-P(35S)(OH)-O-R'] mit 35S markiert. Da die β-Partikel, die von 35S emittiert werden, ungefähr 10-mal weniger Energie besitzen als die von 32P, findet eine geringere radiologische Zerstörung der markierten DNA-Ketten statt und die Banden auf den Autoradiogrammen der Sequenzierungsgele sind schmaler und weniger diffus.
Alternativ werden Farbstoffe verwendet, die bei unterschiedlichen Wellenlängen fluoreszieren. Dabei kann entweder bereits der Primer durch kovalente Bindung des Farbstoffmoleküls an sein 5'-Ende markiert werden oder die 2',3'-Didesoxyribonucleotide durch die kovalente Bindung jeweils unterschiedlicher Farbstoffmoleküle. Die Verwendung dieses farbstoffmarkierten Materials hat die Anwendung von zwei leicht unterschiedlichen DNA-Sequenzierungsprozessen zur Folge: die "Farbstoff-Primer-Markierung" und die "Farbstoff-Terminator-Markierung". Beide Prozesse wurden mittlerweile automatisiert.
Bei der "Farbstoff-Primer-Markierung" werden vier unterschiedlich fluoreszierende Farbstoff-Primer-Spezies verwendet, die dadurch entstehen, dass der gleiche Primer in vier Proben mit einem von vier unterschiedlichen Fluoreszenzfarbstoffen reagiert. Man spricht hier z.B. von blauen, grünen, roten und gelben Primern, obwohl dies in Wirklichkeit nicht den Farbdifferenzen ihrer spektrophotometrisch unterschiedenen Fluoreszenz entspricht. Die vier Inkubationen enthalten einen unterschiedlich "gefärbten" Primer, der mit der DNA, die sequenziert werden soll, hybridisiert ist, sowie die DNA-Polymerase, die vier verschiedenen dNTPs und eine geringe Menge eines der vier ddNTPs. Dadurch wird jedes ddNTP willkürlich einem unterschiedlich "gefärbten" Primer zugeteilt (z.B. ddGTP dem roten Primer, usw.). Jede Inkubationsmischung enthält dann Moleküle, die an ihrem 5'-Ende mit einem Primer einer bestimmten Farbe markiert sind und an ihrem 3'-Ende das 2',3'-Didesoxyribonucleotid, das dem Primer zugeordnet wurde, tragen. Die vier Sätze primermarkierter Ketten werden vereinigt und elektrophoretisch in einer einzigen Bahn eines Polyacrylamid-Sequenzierungsgels getrennt. Die Nucleotidsequenz wird dann aufgrund der "Farbe" (d.h. Farbstofffluoreszenz) der Banden im Elektrophoretogramm bestimmt, wenn die Bahn durch einen spektrofluorometrischen Detektor läuft.
Bei der "Farbstoff-Terminator-Markierung" werden vier unterschiedlich fluoreszierende Farbstoff-ddNTP-Spezies (die durch die Reaktion von ddATP, ddGTP, ddCTP und ddTTP mit jeweils einem anderen Farbstoff synthetisiert werden) in geringen Mengen in demselben Reaktionsgefäß mit dem Primer (der mit der DNA hybridisiert ist, die sequenziert werden soll), den vier dNTPs und der DNA-Polymerase inkubiert. Hierbei entsteht ein Gemisch von vier Sätzen an DNA-Ketten, wobei jede Komponente an ihrem 5'-Ende den Primer und an ihrem 3'-Ende ein unterschiedlich fluoreszierendes 2',3'-Didesoxyribonucleotid trägt. Die Aufarbeitung des Gemischs und die Interpretation der Ergebnisse erfolgt ähnlich wie bei der Methode der Farbstoff-Primer-Markierung.
Als DNA-Polymerase wurde ursprünglich das Klenow-Fragment der DNA-Polymerase I von E. coli verwendet, das jedoch verschiedene Nachteile hat: a) niedrige Progressivität (d.h. das Enzym dissoziiert leicht wieder von der Matrize ab, obgleich die Katalyse noch weiterlaufen könnte), b) schwache Akzeptanz von ddNTPs und c) Intoleranz gegenüber dNTP-Analoga. Sie wurde deshalb großenteils durch die DNA-Polymerase aus thermophilen Bakterien (z.B. Taq-DNA-Polymerase aus Thermus aquaticus) und aus Bakteriophagen T7 ersetzt. Die Taq-DNA-Polymerase besitzt hohe Progressivität, ist tolerant gegenüber ddNTPs und dNTP-Analoga und ist insbesondere nützlich für die Sequenzbestimmung von DNA, deren komplementäre DNA-Stränge bei 37°C extensiv intrasträngige Sekundärstrukturen bilden (da das Enzym aus einem thermophilen Organismus stammt, kann es bei Inkubationstemperaturen von 70-75°C eingesetzt werden, bei denen wasserstoffbrückengebundene Sekundärstrukturen ausgeschlossen sind). Die erfolgreichsten T7-DNA-Polymerasen sind so modifiziert worden, dass die 3'→5'-Exonucleaseaktivität des Enzyms reduziert (z.B. SequenaseTM 1) bzw. sogar eliminiert (z.B. SequenaseTM 2) wurde. Sie besitzen eine hohe Progressivität, eine große Toleranz gegenüber ddNTPs und dNTP-Analoga, eine hohe Elongationsgeschwindigkeit (~300 Nucleotide werden je Sekunde addiert; im Vergleich dazu: Klenow-Fragment 30-45 und Taq-DNA-Polymerase 35-100) und können mit kurzen Primern verwendet werden. Die Taq-DNA-Polymerase und die Sequenasen können dazu eingesetzt werden, in einem einzigen Reaktionsansatz eine DNA-Sequenz zu bestimmen, die mehrere Hundert Nucleotide lang ist.
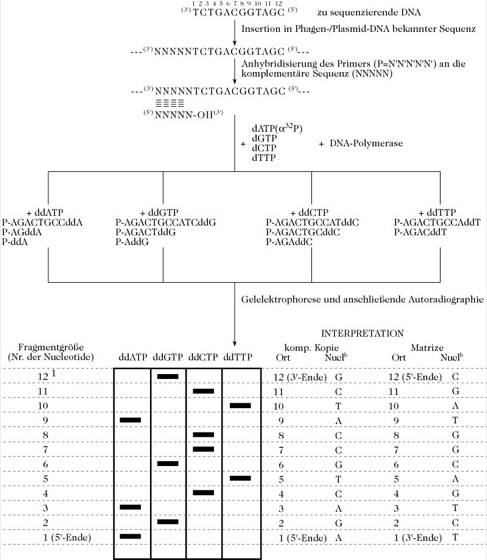
Sanger-Sequenzierung. Das Schema zeigt die Methode angewandt auf die Bestimmung der Nucleotidsequenz eines hypothetischen DNA-Abschnitts. a = Anzahl der Nucleotide in der DNA-Kette, die komplementär zur Ursprungs-DNA ist; die konstante Anzahl der Nucleotide im Primer P, der am 5'-Ende gebunden ist, wird nicht berücksichtigt. b = Nucleotid; komp. = komplementär.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.