Lexikon der Biochemie: Topoisomerasen
Topoisomerasen, Enzyme, die topologische Isomere zirkulärer Doppelstrang-DNA durch Änderung des Grads der Superverdrillung (Superhelizität; Superhelix) ineinander umwandeln. Bei Prokaryonten spielt der Superverdrillungsgrad vermutlich eine wichtige Rolle für die Steuerung der Replikation, Transcription, Rekombination, Integration, Transposition und Renaturierung einzelsträngiger zirkulärer DNA. Außerdem kann die Topoisomerisierung für die chemomechanische Aktivität der DNA bei Prozessen, wie dem Füllen von Phagenköpfen, dem DNA-Transfer im Verlauf bakterieller Konjugation sowie der Mitose wichtig sein. T. katalysieren auch Bildung und Auflösung verknoteter oder als Catenane vorliegender zirkulärer Duplex-DNA.
T. I spalten einen Strang der DNA-Doppelhelix auf, so dass sich die Verknüpfungszahl in Einerschritten ändert. Es wird kein ATP benötigt. Reaktionen, die durch T. I katalysiert werden, kann man sich so vorstellen, dass entweder ein vorübergehend aufgespaltener Strang sich um seinen komplementären Nachbarstrang dreht (Abb.1) oder dass der nicht gespaltene Strang durch den vorübergehenden Bruch wandert. Prokaryontische T. I werden über eine Phosphotyrosinbindung kovalent an das 5'-Ende des Bruchs gebunden. Dadurch wird die Energie der gespaltenen Phosphodiesterbindung konserviert und es den beiden Enden der gespaltenen Stränge nach der Topoisomerisierung ermöglicht, sich wieder zu vereinen. T. I kann nur negativ, jedoch nicht positiv superspiralisierte DNA entspannen und benötigt Mg2+ für ihre Aktivität .
Eukaryontische T. I entspannt sowohl positiv als auch negativ superspiralisierte DNA. Während der Katalyse ist das Enzym mit dem 3'-Ende des Bruchs über eine Phosphotyrosinbindung verknüpft. Rattenleber-T. I katalysiert in vivo die Bildung von Chromatin-ähnlichem Material aus entspannter zirkulärer DNA und Kernhistonen. Das lässt vermuten, dass die eukaryontische T. I bei der Chromatinbildung in vivo eine Rolle spielt. Dies steht im Einklang damit, dass die eukaryontischen Enzyme fast ausschließlich in der Chromatinfraktion gefunden werden. Es wird vermutet, dass Superverdrillung oder Entwindung der DNA innerhalb des Nucleosoms eine erhöhte Spannung auf den Rest des DNA-Moleküls ausüben, das dann durch die Wirkung der T. I entspannt wird. Diese Theorie muss noch mit der Tatsache in Einklang gebracht werden, dass Chromatin in vivo aus neu replizierter, diskontinuierlicher DNA zusammengesetzt sein kann (Chromatin).
Auch folgende, in der Literatur beschriebene Enyzme, zählen zu den T. I: E.-coli-ω-Protein (identisch mit Eco-DNA-T., E.-coli-Swivelase und E.-coli-T. I), entdrillende Enzyme, Einzelstrangbruch-schließende Enzyme, Relaxationsprotein, DNA-entspannendes Enzym.
T. II unterstützen einen Doppelstrangbruch, durch den ein anderer Abschnitt der nichtgespaltenen Doppelhelix durchwandert, bevor der Bruch wieder geschlossen wird (Abb. 2). Die Verknüpfungszahl ändert sich deshalb in Zweierschritten. T. II kann außerdem sowohl aus geschlossener, zirkulärer DNA Catenane bilden und diese auflösen als auch Superverdrillungen entspannen. DNA-Gyrase ist eine T. II, die sich von allen anderen T. darin unterscheidet, dass sie die Verdrehung der zirkulären DNA-Helix erhöhen kann, d.h. sie kann entspannte zirkuläre DNA in eine Superhelix überführen. Hiermit ist eine Erhöhung der Freien Energie verbunden, die durch ATP zur Verfügung gestellt wird. In Abwesenheit von ATP wird superspiralisierte DNA durch DNA-Gyrase entspannt. Die durch Gyrase bewirkte Superverdrillung ist immer negativ, so auch bei intrazellulärer DNA. Folgende Reaktionen werden ebenfalls von Gyrase katalysiert: DNA-abhängige ATP-Hydrolyse, Verknoten und Entknoten sowie Bildung und Auflösung von Catenanen der zirkulären Duplex-DNA.
Gyrasen werden durch zwei Gruppe von Antibiotika inhibiert: 1) Novobiocin, Coumermycin und Clorobiocin; 2) Nalidixinsäure und Oxolinsäure. E.-coli-Mutanten, die gegen eine der beiden Antibiotikagruppen resistent sind, enthalten Antibiotika-resistente DNA-Gyrase. Daher wurden der Locus für die Nalidixinsäure- und Oxolinsäureresistenz (48min auf der E.-coli-Karte) von nal A in gyr A und der Locus für die Resistenz gegen Coumermycin, Novobiocin und Clorobiocin (82min) in gyr B (vorher cou) umbenannt. Alle diese Antibiotika sind Inhibitoren der DNA-Replikation, weshalb angenommen wird, dass DNA-Gyrase an diesem Vorgang beteiligt ist. Gyrase ist ein äquimolarer Komplex aus zwei Proteinen (A und B) und kommt in Lösung vermutlich in Form des A2B2-Komplexes vor. Jedes Protein stellt den Angriffspunkt einer Antibiotikafamilie dar. DNA-Gyrase-Aktivität wurde in keinem eukaryontischen Organismus gefunden.
Andere T. II katalysieren eine ATP-abhängige Entspannung superverdrillter DNA und werden in der Literatur als ATP-abhängige DNA-entspannende Enzyme beschrieben. Der erste Vertreter dieser Gruppe wurde aus mit T4-Phagen infiziertem E. coli isoliert [L.F. Liu et al. Nature281 (1979) 456-461]. Das gereinigte Enzym besteht aus zwei Proteinkomponenten mit Mr von 63kDa und 59kDa. In vivo scheint es für die Initiierung der DNA-Replikationsgabel (jedoch nicht für deren Fortschreiten) verantwortlich zu sein. In vitro entspannt es sowohl negativ als auch positiv superverdrillte DNA. Wenn große Mengen der T4-DNA-T. mit zirkulärer doppelsträngiger DNA in Abwesenheit von ATP inkubiert werden, entstehen verknotete DNA-Moleküle. Diese werden bei Inkubation mit katalytischen Enzymmengen in Gegenwart von ATP in eine einfache zirkuläre Form zurückverwandelt. Ähnliche T. II wurden auch aus vielen verschiedenen eukaryontischen Quellen (z.B. Drosophila-Embryo, Xenopus-laevis-Keimbläschen, Rattenlebermitochondrien, Kalbsthymus, HeLa-Zellkerne, Hefe) isoliert.
Eine frühere Nomenklatur unterschied zwischen T. I und II in Abhängigkeit davon, ob sie positive und negative oder nur negative Superverdrillungen entspannen. Diese ist jedoch nicht weit verbreitet und sollte vermieden werden. Da topologische Änderungen der DNA mit vorübergehendem Bruch und Wiederverbinden der DNA-Stränge verbunden ist, kann jedes Enzym, das DNA-Bruch verursacht, auch T.-Aktivität zeigen. Umgekehrt ist es möglich, dass Enzymen, die zuerst auf Grund ihrer T.-Aktivität gefunden wurden, auch andere physiologische Aufgaben zukommen, an denen eine vorübergehende DNA-Spaltung beteiligt ist.
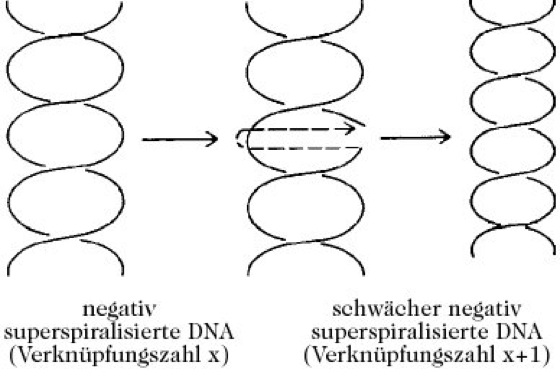
Topoisomerasen. Abb. 1. Ein Modell für die katalytische Aktivität der Topoisomerase I. Ein Einzelstrang der DNA-Doppelhelix wird vorübergehend gespalten. Dieser dreht sich dann vor dem Wiederverknüpfen um den nicht gespaltenen Strang herum.
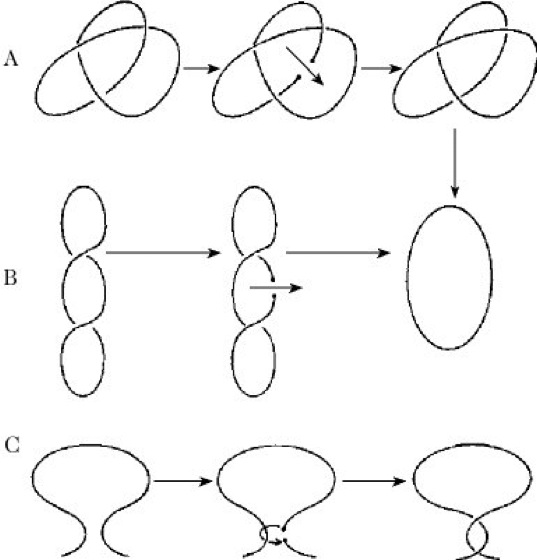
Topoisomerasen. Abb. 2. Reaktionen, die durch Topoisomerasen II katalysiert werden. Die durchgezogenen Linien stellen in allen Fällen doppelsträngige DNA dar. Der vorübergehende Bruch vollzieht sich in beiden Strängen der Doppelhelix. A: Auflösung einer verknoteten zirkulären Duplex. B: Entspannung der Superverdrillung einer zirkulären Duplex. C: Eine Schleife einer zirkulären Duplex, an Hand der die Entstehung einer Superspiralisierung gezeigt wird.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.