Kompaktlexikon der Biologie: DNA-Sequenzierung
DNA-Sequenzierung, Methode zur Bestimmung der Basensequenz eines DNA-Abschnittes, wie z.B. eines in einen Vektor einklonierten DNA-Fragmentes oder eines PCR-Produktes (Polymerasekettenreaktion). Die heute gängigste Methode ist die von F. Sanger entwickelte Didesoxy-Methode (Kettenabbruchmethode), bei der ein radioaktiv oder mit Fluoreszenzfarbstoffen markierter komplementärer DNA-Strang in vitro synthetisiert wird, wobei die Verwendung so genannter Didesoxynucleotide zu einem zufallsmäßigen Kettenabbruch und somit zu unterschiedlich langen DNA-Strängen führt. ( vgl. Abb. )
Als Ausgangspunkt der Neusynthese dient ein sequenzspezifischer Primer, der an die einzelsträngige Matrize anbindet. Eine DNA-Polymerase synthetisiert in vier parallelen Reaktionen die Synthese eines komplementären Stranges, wobei neben den vier 2-Desoxyribonucleotidtriphosphaten (dATP, dTTP, dCTP und dGTP) jeweils ein Didesoxytriphosphat (ddATP, ddTTP, ddCTP und ddGTP) zugefügt wird. Dieses wird wie die anderen eingebaut, allerdings kann die Kettenverlängerung aufgrund der fehlenden 3'-OH-Gruppe nicht erfolgen. Auf diese Weise erhält man ein Gemisch mit unterschiedlich langen DNA-Molekülen, an deren 3'-Ende sich stets eine bestimmte Base befindet. Wurde dem Reaktionsgemisch z.B. ddATP beigemischt, enden alle Moleküle mit einem Adenin. Alle vier Parallelansätze werden anschließend in einem denaturierenden Polyacrylamidgel elektrophoretisch nebeneinander aufgetrennt (Elektrophorese), sodass Fragmente, die sich in bezug auf ihre Länge nur um ein Basenpaar unterscheiden, voneinander getrennt werden können. Durch die radioaktive Markierung einer Base mit 32P oder 35S, können die aufgetrennten Fragmente auf einem Röntgenfilm sichtbar gemacht und die Sequenz ermittelt werden.
Diese ursprüngliche Versuchsanordnung der D.-S. hat inzwischen zahlreiche Modifikationen erfahren. Anstelle des ursprünglich verwendeten Klenow-Fragmentes, das nur kurze DNA-Stränge synthetisieren kann und dem GC-reiche Sequenzbereiche Schwierigkeiten bereiten, wird heute eine modifizierte DNA-Polymerase des Bakteriophagen T7 verwendet. Auch die Taq-Polymerase kann zur D. – S. eingesetzt werden. Anstelle der radioaktiven Markierung tritt heute die Verwendung fluoreszierender Verbindungen, die im Zuge der Automatisierung der D. – S. eine Auswertung mittels eines DNA-Sequenzers gestatten. Auf diese Weise können DNA-Sequenzen direkt ausgewertet werden.
Die zur D. – S. früher häufig verwendete Maxam-Gilbert-Methode wird heute nur noch selten und für besondere Zwecke (z.B. DNaseI-Footprinting) benutzt. Sie beruht auf chemisch induzierten basenspezifischen Strangbrüchen und anschließender Auftrennung der entstandenden Fragmente, die in analoger Weise zur oben beschriebenen Didesoxy-Methode ausgewertet werden können.
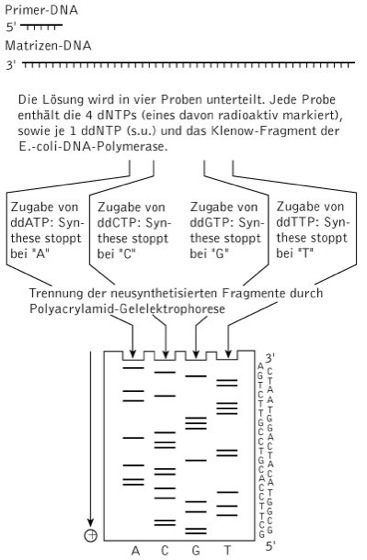
DNA-Sequenzierung: DNA-Sequenzierung nach Sanger
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.