Kompaktlexikon der Biologie: Restriktionsendonucleasen
Restriktionsendonucleasen, Restriktionsenzyme, Typ der Endonucleasen, die doppelsträngige DNA sequenzspezifisch hydrolysieren („schneiden“). R. sind an der so genannten Restriktion beteiligt. Ihre Entdeckung und die damit einhergehende Entdeckung der R. als deren molekulare Ursache in den 1950er bis 1970er-Jahren konnte zur Beilegung des Streits zwischen Lamarckisten und Genetikern beitragen, die die Beobachtung, dass sich Phagen nach einem Transfer auf andere Bakterienstämme zunächst schlecht, dann aber plötzlich stark vermehren, auf unterschiedliche Weise interpretierten. Für ihre Arbeiten zu den R. erhielten W. Arber, D. Nathans und H.O. Smith im Jahr 1978 den Nobelpreis für Physiologie oder Medizin.
Von den drei Klassen, in die sich R. einteilen lassen, sind die R. der Klasse II von großer Bedeutung, weil sie sich in ihrer Eigenschaft als „molekulare Scheren“ für molekularbiologische Arbeitstechniken verwenden lassen. Diese R. erkennen spezifisch vier bis acht Basenpaare umfassende Sequenzmotive, die i.d.R. eine zweiachsige Symmetrieebene aufweisen (Palindrom). Die hydrolytische Spaltung der Phosphodiesterbindung kann dabei entweder innerhalb der palindromischen Erkennungssequenz oder unmittelbar in deren Nähe erfolgen. Dabei können je nach Enzym DNA-Fragmente mit doppelsträngigen glatten Enden (blunt ends) entstehen, oder aber Fragmente gebildet werden, deren 3'- oder 5'-Enden einzelsträngig sind. Sie werden als klebrige Enden (sticky ends) bezeichnet. Die R. der beiden anderen Klassen schneiden die DNA bis zu 1000 bp weit entfernt von der Erkennungssequenz.
Die Nomenklatur der R. setzt sich aus dem Organismus und dem jeweiligen Stamm zusammen, der ursprünglich zur Isolierung eines R. verwendet wurde. Römische Zahlen weisen zudem auf die Reihenfolge der Isolierung hin. Eco RI bedeutet somit, dass dieses R. aus Escherichia coli und dem Stamm RY 13 als erstes isoliert wurde. Die R. einer Reihe unterschiedlicher Mikroorganismen erkennen dieselbe Sequenz, sie werden als Isoschizomere bezeichnet.
Für den Einsatz im Laboralltag werden über hundert verschiedene R. heute i.d.R. mit gentechnischen Verfahren im großen Maßstab produziert. ( vgl. Tab. )
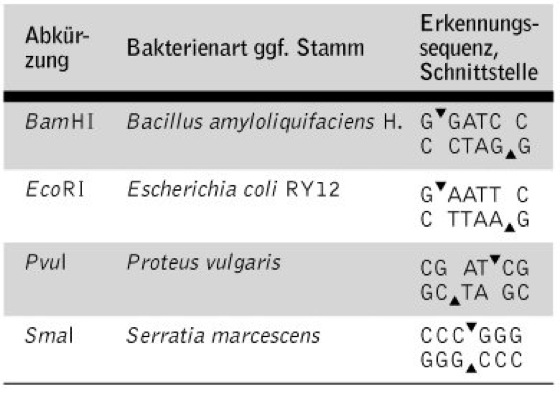
Restriktionsendonucleasen: Auswahl repräsentativer Restriktionsenzyme, die sechs Nucleotidpaare als Erkennungssequenz nutzen; BamHI und EcoRI erzeugen dabei sticky ends mit 5'-Überhängen, PvuI solche mit 3'-Überhängen und SmaI erzeugt blunt ends
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.