Lexikon der Biologie: Expressionsbibliothek
Expressionsbibliothek, Expressionsbank, besondere Art einer Genbibliothek (Genbank), bei der fragmentierte genomische DNA oder cDNAs eines Organismus in geeignete Expressionsvektoren einkloniert werden, um für Proteine codierende Sequenzen exprimieren und identifizieren zu können. Bei einer repräsentativen Expressionsbibliothek muß ebenso wie bei einer normalen Genbibliothek gewährleistet sein, daß das gesamte Genom bzw. der gesamte mRNA-Pool, repräsentiert durch die korrespondierenden cDNAs, in der Expressionsbibliothek enthalten ist. Expressionsbibliotheken können in geeigneten Plasmid- oder Phagen-Vektoren angelegt werden. Bevorzugt werden im allgemeinen jedoch Phagen-Derivate (Bakteriophagen), da mit diesen die Effizienz der Transformation (Zelltransformation) der Wirtszellen (meist für die Expression fremder Proteine besonders geeignete Stämme von Escherichia coli) höher ist als mit Plasmiden. Expressionsbibliotheken prokaryotischer Organismen können hierbei als genomische oder cDNA-Bibliotheken (subtraktive cDNA-Bibliothek) angelegt werden. Soll jedoch eine von einem höheren eukaryotischen Organismus vorliegende Expressionsbibliothek in Escherichia coli-Stämmen zur Expression gebracht werden, sollte hierbei von einer cDNA-Bibliothek ausgegangen werden, da in dieser, bedingt durch die Vorgehensweise bei der Herstellung der cDNA, keine Intronen enthalten sind, wie es in einer genomischen Bibliothek höherer Eukaryoten der Fall ist. Die Intronen könnten von den Wirtsbakterien aus den entstehenden mRNAs nicht durch Spleißen entfernt werden und würden so häufig zu unbrauchbaren Translationsprodukten (Translation) führen. Bei der Konstruktion von Expressionsbibliotheken werden die in die Vektoren einklonierten Fragmente codierender Sequenzen im allgemeinen an Stellen eingefügt, die eine induzierbare Expression als Fusionsprotein (Fusionsgen) erlauben. Dies ist insbesondere bei der Verwendung von cDNAs wichtig, da diese keine eigenen Promotoren besitzen, von denen aus die korrespondierenden mRNAs transkribiert werden könnten. Auch würden eukaryotische Promotoren und Ribosomen-Bindestellen bei Expression in prokaryotischen Systemen von diesen nicht erkannt werden. Bewährt hat sich eine Fusion mit dem Gen für die β-Galactosidase des Lactose-Operons aus Escherichia coli, lacZ ( vgl. Abb. ). Die Fragmente werden über eine singuläre Restriktionsstelle in den Lese-Raster des lacZ-Gens inseriert. Ein Sechstel aller inserts wird in der richtigen Orientierung und im richtigen Leseraster mit dem lacZ-Gen liegen und so nach Induktion des lacZ-Promotors durch einen Induktor zusammen mit dem vorangeschalteten Anteil der β-Galactosidase als Fusionsprotein exprimiert. Dieses System findet z.B. in den von dem Lambda-Phagen abgeleiteten Expressionsvektoren λgt11 oder λZAP Anwendung. Es hat außerdem den Vorteil, daß unter Zugabe einer farblosen Indikatorsubstanz (X-Gal) nur diejenigen Plaques blau erscheinen, die durch Phagen hervorgerufen wurden, welche kein insert in ihrem lacZ-Leseraster besitzen. Nur diese exprimieren nämlich eine funktionsfähige β-Galactosidase, welche die Indikatorsubstanz zu blauen Spaltprodukten umsetzen kann. Bei insert-tragenden Phagen führt die geänderte, neue codierende Sequenz zu einem enzymatisch inaktiven Fusionsprotein und somit zu hellen Phagenplaques. Dieser Farbtest ermöglicht eine Bestimmung des Titers der inserts tragenden Phagen, die schließlich zur Expression von Fusionsproteinen führen können. Die Identifizierung eines bestimmten Fusionsproteins und damit eines bestimmten rekombinanten insert tragenden Phagenklons wird häufig durch ein Screening mit spezifischen Antikörpern gegen das Protein durchgeführt, dessen zugrundeliegende codierende DNA-Sequenz man ermitteln will. Der radioaktiv markierte oder über einen Sekundärantikörper sichtbar zu machende primäre Antikörper bindet spezifisch an diejenigen nach Induktion exprimierten Fusionsproteine, die Teile des gesuchten Proteins enthalten, und identifiziert somit die entsprechenden Klone. Für die Suche nach Klonen mit codierenden Sequenzen für DNA-bindende Proteine kann die Methode des recognition site screening eingesetzt werden. Bei der Suche nach codierenden Sequenzen für Proteine mit enzymatischer Aktivität kann es in seltenen Fällen vorkommen, daß das Fusionsprotein die enzymatisch aktive Domäne beinhaltet. Hier kann der Klon zum Teil direkt durch die Enzymaktivität, im Idealfall durch eine Farbreaktion nachgewiesen werden.
E.G.
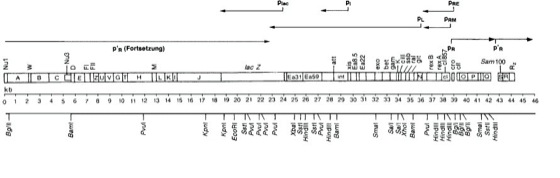
Expressionsbibliothek
Zur Konstruktion einer Expressionsbibliothek in dem Expressionsvektor λgt11 wurden nicht-essentielle Teile des Genoms des Lambda-Phagen durch das lacZ-Gen aus Escherichia coli ersetzt. Werden cDNAs, die mit Linker-DNA für das Restriktionsenzym EcoRI versehen wurden, in die singuläre EcoRI-Schnittstelle innerhalb des lacZ-Gens dieses Vektors einkloniert, so wird ein Sechstel der Inserts in richtiger Orientierung und im richtigen Leseraster vorliegen. Nach Infektion geeigneter Wirtsbakterien wird durch die Induktion des lac-Promotors plac die Expression von Fusionsproteinen initiiert, über die die Phagenklone und nachfolgend die zugrundeliegenden codierenden DNA-Sequenzen identifiziert werden können.
Unterste Linie: Positionen für Schnittstellen der angegebenen Restriktionsenzyme; kb Längenmarkierung in Kilobasenpaaren; Boxen: Phagengene mit Bezeichnungen; mittlere Box: lacZ-Gen, codierend für das Enzym β-Galactosidase aus E. coli; px: Lokalisation verschiedener Promotoren innerhalb des Phagengenoms und von diesen ausgehende Transkripte (Pfeile).
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.