Lexikon der Biologie: Ribonucleinsäuren
Ribonucleinsäuren, Abk. RNS und RNA (engl.), hochpolymere Kettenmoleküle (Biopolymere), in denen als monomere Bausteine vorwiegend die 4 Standard-Ribonucleosidmonophosphate Adenosin-5'-monophosphat (AMP, Adenosinmonophosphat), Cytidin-5'-monophosphat (CMP, Cytidinmonophosphate), Guanosin-5'-monophosphat (GMP, Guanosinmonophosphate) und Uridin-5'-monophosphat (UMP, Uridinmonophosphat) in gebundener Form enthalten sind. Durch Veresterung der 5'-Phosphatgruppe jedes Grundbausteins mit der 3'-Hydroxylgruppe des jeweils benachbarten Monomeren bilden sich die unverzweigten RNA-Ketten ( ä vgl. Abb. ). Unter physiologischen Bedingungen liegt RNA ( ä vgl. Infobox ) nicht in der Säureform, sondern als Poly-Anion mit je einer negativen elektrischen Ladung pro Nucleotidrest vor. Die zur Elektroneutralität erforderlichen Kationen sind sowohl einfache anorganische Kationen (Na+, K+, NH4+) als auch Amine oder basische Proteine. – RNA wird funktionell in 3 Hauptklassen unterteilt: Boten-RNA (engl. messenger-RNA; Abk. mRNA), ribosomale RNA (Abk. rRNA) und transfer-RNA (Abk. tRNA). Daneben existieren noch sog. kleine RNAs (snRNA und scRNA, s.u.). Die 3 Hauptklassen sind an der Translation der genetischen Information wesentlich beteiligt und kommen daher in allen lebenden Zellen bzw. Organismen vor. Die Synthese dieser 3 RNA-Klassen erfolgt durch Transkription der entsprechenden Gene und anschließende Reifung der primären Transkripte durch Prozessierung (u.a. Capping, Polyadenylierung, RNA-Editing, Entfernen intercistronischer Bereiche; spleißen). Bei bestimmten Viren (RNA-Viren, RNA-Tumorviren), darunter Bakteriophagen (einzelsträngige RNA-Phagen), ist RNA Träger der genetischen Information. Bei diesen erfolgt die RNA-Synthese durch Replikation (RNA-Replikation im Gegensatz zu DNA-Replikation). Ferner sind die aus kurzkettigen, ringförmigen RNAs aufgebauten Viroide zu nennen. Zelluläre RNA kommt sowohl im Zellkern, dem Ort der RNA-Synthese (hn-RNA) und der RNA-Prozessierung, als auch im Cytoplasma, dem Ort, in dem RNAs ihre Funktion beim Translationsprozeß ausüben, vor. Darüber hinaus enthalten auch die genetisch semiautonomen Organellen, die Mitochondrien und Chloroplasten, eigene RNAs (mRNA, tRNA und rRNA), die von den jeweiligen Organellen-Genomen (Chondrom, Plastom) codiert werden. – Die Stabilität von RNAs ist sehr unterschiedlich. Während mRNA meist kurzlebig ist und z.B. in Bakterien Halbwertszeiten im Minutenbereich zeigt, sind tRNA und rRNA meist über viele Zellgenerationen stabil. Aus der kurzen Lebensdauer von mRNA ergibt sich ein energetisch hoher Preis für die fortlaufende Bildung von mRNA-Molekülen als Informationsüberträger (Energiekonservierung). Andererseits ist die Kurzlebigkeit die Basis für die Steuerung von Genaktivitäten (Genaktivierung [Farbtafel]) und begründet die Fähigkeit zur schnellen genregulatorischen Antwort (Genregulation [Farbtafel]) auf Veränderung der Umwelt oder bei der Steuerung von Entwicklungsprozessen. Der Abbau von RNA sowie die Spaltung von Primärtranskripten zur Freisetzung von tRNAs und rRNAs und manchen mRNAs erfolgt durch Ribonucleasen. – Neben den 4 Standard-Nucleotiden sind besonders in tRNAs, aber auch in rRNAs, Nucleotide mit modifizierten Basen oder mit 2'-O-methylierten (Methylierung) Riboseresten enthalten. Eukaryotische mRNAs besitzen an den 5'-Enden eine 7-Methylguanosin-Gruppe (Methylguanosin) als modifizierte Base (Capping). Mit Ausnahme der RNA einiger Viren sind RNA-Moleküle einzelsträngig. Ohne den fehlenden Gegenstrang können sie keine basengepaarte Doppelhelix (Desoxyribonucleinsäuren) mit proportionierten Basenverhältnissen ausbilden (Basenzusammensetzung). Dennoch gibt es auch in RNA-Ketten Bereiche, in denen durch Zurückfaltung und intramolekulare Basenpaarungen ein Doppelstrang entsteht, dessen Stabilität jedoch häufig durch Fehlpaarungen und/oder die Kürze der Bereiche reduziert ist. Die gepaarten Bereiche (Stamm-Strukturen) besitzen jeweils an einem Ende kürzere ungepaarte Schleifen. Solche Stamm-Schleifen-Strukturen (Haarnadelschleifen), die wegen ihrer Entstehung durch Wasserstoffbrücken zwischen gepaarten Basen als Sekundärstrukturen bezeichnet werden, kommen besonders bei tRNA (Kleeblattstruktur;transfer-RNA [Abb.]) und rRNA (Ribosomen [Abb.]) vor. Im Gegensatz zur durchgehenden Doppelhelix-Struktur von DNA, die sich zwischen zwei Einzelsträngen ausbildet, bestehen RNA-Sekundärstrukturen aus mehreren (tRNA) oder sogar sehr vielen (rRNA), kürzeren Doppelstrangbereichen, die durch Einzelstrangbereiche voneinander getrennt sind und die sich durch Zurückfaltung innerhalb einer Kette bilden. Bei tRNA ist die Faltung der Sekundärstruktur zu einer räumlichen Konformation, zur Tertiärstruktur, bekannt. Noch komplexere Tertiärstrukturen nehmen rRNAs in Wechselwirkung mit Proteinuntereinheiten innerhalb von Ribosomen ein. mRNA besitzt praktisch keine übergeordneten Sekundärstrukturen. Bei ihrer Funktion als Vermittler der genetischen Information beim Translationsprozeß wäre eine Faltung und die dadurch bedingte Unzugänglichkeit von Signalstrukturen oder Aminosäure-Codonen hinderlich. Ausnahmen von dieser Regel sind z.B. Stamm-Schleifen-Strukturen von Bereichen, die als Attenuatoren bzw. Terminatoren fungieren. – In der Zelle kommt RNA praktisch nicht in freier Form vor. Vielmehr ist sie mit unterschiedlichen Proteinen (RNA-bindende Proteine) zu Ribonucleoprotein-Partikeln (Ribonucleoproteine, Abk. RNP) assoziiert. Die stabilsten und zahlreichsten RNPs sind die Ribosomen bzw. Polyribosomen. In den Zellkernen eukaryotischer Zellen (Eucyte, Zelle [Tab.]) wird mRNA schon während der Transkription mit Proteinen beladen und bildet so die zu 80% aus Protein bestehenden hnRNP-Partikel (auch nucleäre Informatorkomplexe genannt; hn-RNA). Sie enthalten neben den sehr fest gebundenen, sog. Core-Proteinen, die etwa 75% des Proteinanteils ausmachen, auch Enzymproteine, wie RNA-Prozessierungsenzyme (Capping-Enzym, RNasen, poly(A)-Polymerase) und Proteinkinasen. Beim Durchtritt reifer mRNA durch die Kernporenkomplexe werden die Proteine ausgetauscht und cytoplasmatische mRNP-Partikel (Informosomen genannt) gebildet (messenger-Ribonucleoproteine). Sie enthalten Subpartikel (scRNP) mit kleinen cytoplasmatischen RNAs (small cytoplasmatic RNA, scRNA). Auch im Zellkern existieren kleine RNAs (small nuclear RNAs, Abk. snRNAs), die mit Proteinen zu snRNP-Partikeln (small nuclear RNP) vereinigt sind. Die zu dieser Klasse gehörende, aus 165 Nucleotiden aufgebaute sog. U1snRNA (UsnRNAs, UsnRNPs) bewirkt das korrekte Zusammenführen von Exon-Grenzen (Exon-Intron-Grenze) beim Spleißen von RNA (Genmosaikstruktur [Abb.]). – Neben den bereits genannten können RNAs weitere Funktionen ausüben. Bei Bakterien und Niederen Tieren werden antisense-RNAs gefunden, die regulatorische Funktion bei verschiedenen Prozessen ausüben. Manche RNAs zeigen allein (z.B. bei 26S-rRNA von Tetrahymena [Hymenostomata]) oder komplexiert mit Proteinen (z.B. bei RNAse P) katalytische Eigenschaften. Solche erst seit etwa 20 Jahren als Ribozyme bekannten RNAs, die Aufgaben übernehmen, welche sonst nur von Enzymen wahrgenommen werden, sind ebenso wie die durch ihre spezifischen Bindungseigenschaften ausgezeichneten Aptamere vor allem aus pharmakologischer Sicht interessant und können z.B. mittels in-vitro-Evolution erzeugt bzw. selektioniert werden. – In den letzten Jahren zeigt sich in zunehmendem Maße, daß RNAs im Zellgeschehen weit mehr Funktionen zukommen als ihre schon lange bekannte Vermittlerrolle bei der Umsetzung des genetischen Codes bzw. ihre katalytischen Aufgaben. So üben zahlreiche neuentdeckte, winzige, meist nur 21–22 (maximal 30) Nucleotide große RNAs bei Entwicklungsvorgängen und anderen biologischen Prozessen eine lange Zeit unterschätzte Steuerungsfunktion bei der Regulation der Genaktivität aus. Solche zunächst bei Caenorhabditis elegans entdeckten regulatorischen „Mini“-RNAs, auch als mikro-RNAs (micro-RNAs, Abk. miRNAs) bezeichnet, kennt man inzwischen bei nahezu allen Organismen. Ebenso klein sind die jüngst entdeckten, sog. Interferenz-RNAs (kleine interferierende RNAs). Beide Mini-Moleküle (mikro- und Interferenz-RNAs) werden von speziellen, Dicer genannten Enzymen aus größeren, zu einem Doppelstrang zusammengelagerten Molekülen zurechtgeschnitten: sie bestehen aus einzelsträngigen RNA-Molekülen, die haarnadelförmig zu Doppelstrangbereichen aufgefaltet sind. Während die mikro-RNA aus zelleigener RNA hervorgeht, entstammt die Interferenz-RNA einer fremden, „illegal“ in die Zelle eingewanderten RNA (z.B. von RNA-Viren). Beide Moleküle lagern sich nach dem „Zurechtschneiden“ an komplementäre Sequenzen (Komplementarität) an – bei der mikro-RNA sind dies zelleigene Kopien –, so daß die Translation (Proteinsynthese) gestoppt wird. Die Interferenz-RNA lagert sich an die fremden RNAs an und leitet damit deren Abbau ein. So hemmt sie bei Pflanzen die Vermehrung von RNA-Viren (Pflanzenviren), indem sie für den enzymatischen Abbau des viralen Erbmaterials sorgt. Damit schützt sich die Zelle mit der Interferenz-RNA vor der Zerstörung durch doppelsträngige RNA-Viren und andere intrazelluläre Erreger. Neuerdings mehren sich die Hinweise, daß mikro-RNAs auch eine Kontrollfunktion bei der Transkription, also der Bildung der messenger-RNA, zukommt und sie massive Umlagerungen im Genom bewerkstelligen. Es ist zu erwarten, daß in den nächsten Jahren weitere Facetten in der mittlerweile breiten Palette der Funktionen von RNAs entdeckt werden. Biochemie (Geschichte der), Levene (P.A.T.), RNA-Mapping, RNA-Polymerase, RNA-Replikase, RNA-Topologie, RNA-Welt; RNP-Welt, Transkription – Translation.
H.K./R.S./M.B.
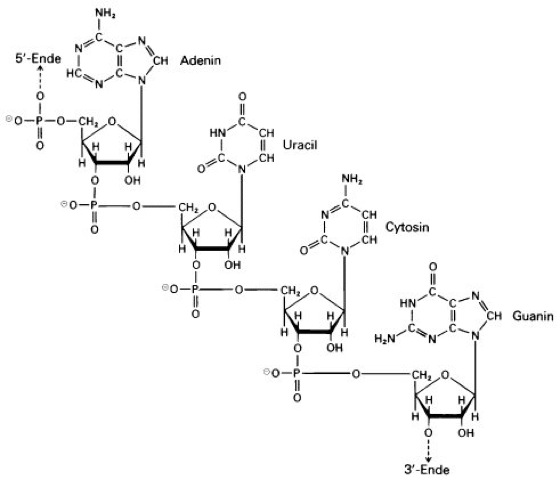
Ribonucleinsäuren
Ausschnitt aus dem Einzelstrang einer Ribonucleinsäure (RNA).
Der Ausschnitt aus der Strukturformel zeigt die Tetranucleotidsequenz AUCG (in der konventionellen 5' → 3'-Richtung, d.h. vom 5´-Ende zum 3'-Ende gelesen). Man beachte, daß die waagerechten P–O–CH2-Bindungen aus Gründen der räumlichen Darstellung erheblich überdehnt sind und in Wirklichkeit gleich lang wie die senkrechten P–O-Bindungen sind. Die Länge der einzelnen Typen von RNA-Molekülen ist sehr unterschiedlich. Kleine RNA-Moleküle sind die transfer-RNAs, die einheitlich eine Länge von etwa 80 Nucleotiden haben. Nicht viel größer sind die kleinen ribosomalen RNAs mit einer Länge von 80–130 Nucleotiden. Danach kommen die verschieden großen ribosomalen RNAs mit Längen von 1500 bis 4000 Nucleotiden. Am längsten, aber auch am unterschiedlichsten lang innerhalb ihrer Gruppe sind die messenger-RNAs mit Längen von bis zu 10.000 Nucleotiden.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.