Lexikon der Biologie: transfer-RNA
transfer-RNAw, transfer-RNS, transfer-Ribonucleinsäure, Abk. tRNA (t-RNA), tRNS (t-RNS), veraltete Bezeichnungen: sRNA (von engl. soluble RNA), lösliche RNA, Akzeptor-RNA, Transport-RNA, eine Gruppe von ubiquitär vorkommenden, relativ kurzkettigen (zwischen 73 und 95 Nucleotidreste; mittlere relative Molekülmasse 25 kDa) RNA-Molekülen (Ribonucleinsäuren), durch die bei der Proteinsynthese (Translation; Proteine) die einzelnen Aminosäuren gebunden (und gleichzeitig aktiviert; Aminosäureaktivierung) und an den mRNA-Ribosomen-Komplex herangeführt werden, um anschließend auf die wachsenden Peptidketten (Peptide, Peptidbindung) transferiert (Bezeichnung!) zu werden (Ribosomen [Farbtafel], Translation). Zwischenprodukte sind dabei Aminoacyl-tRNA und Peptidyl-tRNA (Peptidyl-Transferase [Abb.]), für die jeweils eigene Bindestellen (Bindereaktion) am Ribosom existieren (A-Bindungsstelle, P-Bindungsstelle). Für jede der 20 proteinogenen Aminosäuren existiert mindestens 1, in der Regel jedoch mehrere (bis zu 6) tRNA-Spezies (Isoakzeptoren), die sich sowohl durch die Primärstrukturen als auch die Modifikationen (Basenmodifikation, modifizierte Basen) einzelner Nucleinsäurebasen voneinander unterscheiden. – Im Unterschied zu den anderen beiden RNA-Spezies (messenger-RNA, ribosomale RNA) kommen bei tRNAs zahlreiche Modifikationen vor, die auf Methylierung (Guanosin → 7-Methylguanosin), Desaminierung (Guanin → Inosin), Schwefelsubstitution (Uridin → 4-Thiouridin), Basen-Umlagerungen (Uridin → Pseudouridin) und auf die Sättigung vorhandener Doppelbindungen (Uridin → Dihydrouridin) zurückzuführen sind. Da Organellen- und Artspezifität zur weiteren Multiplizität der tRNA beitragen, dürfte die wahre Zahl der tRNA-Spezies in einer Zelle bei 50 bis 70 liegen. Zur genaueren Unterscheidung sind Kurzbezeichnungen üblich, z.B. bedeutet ![]()
die für Valin spezifische tRNA-Spezies aus Hefe. – Die Aminoacylierung oder Beladung der tRNAs (Kopplung der einzelnen Aminosäuren an ihre jeweiligen tRNA-Spezies) erfolgt unter Energieverbrauch (ATP-Spaltung; Adenosintriphosphat [Abb.]) durch die Bildung einer kovalenten chemischen Bindung zwischen der Carboxylgruppe am C-Terminus (Carboxylterminus) der Aminosäure und einer OH-Gruppe (Hydroxylgruppe) am 3'-Ende der tRNA ( vgl. Abb. 1 ). Diese Reaktion wird durch Aminoacyl-tRNA-Synthetasen katalysiert, wobei für jede Aminosäure ein eigenes Enzym existiert; Isoakzeptoren werden somit durch dasselbe Enzym beladen. Die Enzyme stellen eine heterogene Gruppe dar, deren Reaktion sich wie folgt beschreiben läßt: Aminosäure + ATP + tRNA → Aminoacyl-tRNA + AMP + PPi. Dabei entsteht zunächst ein Anhydrid zwischen der Aminosäure und dem AMP (Adenosinmonophosphat). Die so aktivierte Aminosäure (Aminosäureaktivierung, Aminoacyl-Adenylsäuren) wird anschließend auf die OH-Gruppe der Ribose am 3'-Ende der tRNA übertragen (Esterbildung; Ester). Die dabei entstehende energiereiche Bindung (energiereiche Verbindungen) wird später während der Translation zur Bildung der Peptidbindung zwischen der Aminosäure und der wachsenden Polypeptidkette genutzt. Aminoacyl-tRNA-Synthetasen lassen sich in 2 Klassen einteilen, wobei Klasse I die Aminosäuren mit der 2'-OH-Gruppe der Ribose am 3'-Ende verknüpft, Enzyme der Klasse II hingegen die Veresterung mit der 3'-OH-Gruppe katalysieren. Jede Aminoacyl-tRNA-Synthetase erkennt ihre tRNAs am Anticodon und an weiteren „Identitätsmerkmalen“, d.h., sie geht hochspezifische Wechselwirkungen mit einer kleinen Zahl von speziellen Nucleotiden ein. Diese sehr exakte Zuordnung von tRNAs und entsprechenden Synthetasen wird auch als „zweiter genetischer Code“ bezeichnet. Für die Genauigkeit der Proteinbiosynthese ist die Beladung der tRNAs mit der richtigen Aminosäure von entscheidender Bedeutung, denn bei der Translation selbst ist nur noch das Anticodon der tRNA, nicht mehr die an der tRNA hängende Aminosäure ausschlaggebend. – Die Synthese von tRNAs erfolgt durch Transkription von tRNA-codierenden Genen (tRNA-Gene, tDNA) durch die RNA-Polymerase III (RNA-Polymerase), wobei zunächst längerkettige Vorläufer-tRNAs entstehen (Primärtranskript). Durch Prozessierung werden diese zu den Kettenlängen der reifen tRNAs verkürzt, wobei sowohl an den beiden Enden überschüssige Kettenabschnitte entfernt werden als auch im Falle gespaltener tRNA-Gene ein Spleißen im Innern der Kette erfolgen kann. Gleichzeitig oder sofort anschließend erfahren zahlreiche Basen spezifische Modifikationsreaktionen, die wie bei tRNA aus Hefe bis zu 20% der in tRNA enthaltenen Basen betreffen können (Alanin-tRNA [Abb.]). In geringerem Umfang werden auch einzelne Ribosereste durch 2'-O-Methylierung modifiziert. Die am 3'-Ende jeder tRNA vorkommende, zur Aminosäure-Kopplung essentielle Sequenz CCA (Aminoacyl-tRNA [Abb.]) ist vielfach nicht in den jeweiligen tRNA-Genen codiert. Sie wird in diesen Fällen im Zuge von Reifungsprozessen durch ein spezielles Enyzm (tRNA-CCA-Pyrophosphorylase) nachträglich angehängt. Schließlich werden die durch Gene des Zellkerns codierten reifen tRNAs in das Cytosol transportiert, wo sie als Komponenten der Proteinsynthese wirken. Die genetisch semiautonomen Organellen, Mitochondrien und Chloroplasten, besitzen jeweils einen eigenen, vollständigen Satz von tRNAs, der sich von den cytosolischen tRNAs unterscheidet und der in mitochondrialer DNA bzw. Plastiden-DNA codiert wird. – Die Faltung der tRNA-Primärstrukturen zu den Sekundärstrukturen (= Kleeblattstrukturen) und Tertiärstrukturen (RNA-Topologie) erfolgt sowohl durch intramolekulare Basenpaarungen als auch durch zusätzliche Wasserstoffbrücken und durch Aneinanderstapeln gepaarter Bereiche (Stapelkräfte). – Die Kleeblattstruktur ( vgl. Abb. 2/1 ) gliedert sich in bestimmte Domänen, die unterschiedliche Funktionen wahrnehmen. So ist der aus den beiden Enden bestehende, 7 Basenpaare umfassende Bereich, der als Akzeptor-Stamm, A-Stamm oder Akzeptor-Arm bezeichnet wird, für die Anbindung der jeweiligen Aminosäure verantwortlich. Der Anticodon-Arm (AC-Arm) enthält die für die Translation erforderliche Information, wohingegen die anderen beiden Arme aufgrund des Vorhandenseins modifzierter Nucleotide als DHU-Arm (D-Arm; Dihydrouracil) und TψC-Arm (T-Arm; Pseudouracil) bezeichnet werden ( vgl. Abb. 3 ). Sie dienen vor allem der Stabilisierung der tRNA-Moleküle. Die Tertiärstruktur der tRNAs wurde durch Röntgenstrukturanalyse bestimmt und wird als sog. L-Form bezeichnet ( vgl. Abb. 2/2 ). – Die aus 3 benachbarten Nucleotiden bestehenden Anticodonen jeder tRNA paaren am Ribosom während der Translation mit den entsprechenden Codonen von messenger-RNA (sog. Codon-Anticodon-Paarung). Diese Wechselwirkung ist essentiell für die korrekte Übersetzung der Nucleotidsequenz von mRNA in die Aminosäuresequenz der Proteine. Die Codon-Anticodon-Paarung erfolgt nur für die Positionen 1 und 2 des Anticodons nach den klassischen Basenpaarungsregeln (Basenpaarung [Abb.]). In der 3. Position sind nach der von F. Crick 1965 aufgestellten, sog. Wobble-Hypothese auch nichtklassische Basenpaarungen (G–U, U–G, Inosin–U, Inosin–C, Inosin–A) zulässig, wodurch einzelne tRNAs in der Lage sind, mit mehr als nur 1 Codon zu paaren. So kann das in einer Phenylalanin-spezifischen tRNA enthaltene Anticodon AAG (G in der 3. Position) nicht nur mit dem Codon UUC (klassische Paarung), sondern auch mit dem Codon UUU (G–U-Paar in der 3. Position) paaren, weshalb die beiden für Phenylalanin codierenden Basentripletts (UUU und UUC) allein von dieser tRNA translatiert werden können. Aufgrund dieses Einsparungsprinzips braucht die Zelle nicht für alle 61 Aminosäure-Codonen (genetischer Code [Tab.]) eine eigene tRNA-Spezies, so daß je nach Art des Wobble-Musters und der Codonhäufigkeiten 30–40 tRNA-Spezies ausreichen. Zur Kennzeichnung einzelner tRNA-Spezies werden die Aminosäuren und Anticodonen als Indizes angegeben; z.B. ist eine ![]()
eine Valin bindende tRNA mit dem Anticodon UAC (hier U in der 3. Position), die entsprechend der Wobble-Hypothese mit den beiden Valin-Codonen GUA und GUG paaren kann. – Das Vorhandensein von tRNAs wurde in den 1950er Jahren erstmals von F. Crick vermutet. In den 1960er-Jahren gelang es R.W. Holley, die vollständige Sequenz einer tRNA zu ermitteln. – tRNAs können auch noch ganz andere Funktionen haben. So startet in Prokaryoten und Chloroplasten die Synthese einer Vorstufe der Porphyrinringe (δ-Aminolävulinsäure) mit Glutamyl-tRNAGlu. Bei Retroviren dient eine tRNA als Startermolekül für die reverse Transkriptase bei der RNA-abhängigen DNA-Synthese in der Wirtszelle. Adaptorhypothese, Biochemie (Geschichte der), Cysteinyl-tRNA, Holley (R.W.), Khorana (H.G.); – Transkription – Translation .
H.K./S.Kl.
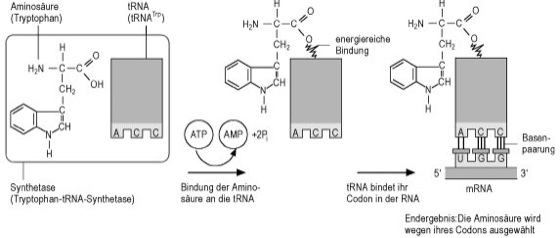
transfer-RNA
Abb. 1: Aminoacylierung („Beladung“) einer transfer-RNA mit einer Aminosäure am Beispiel von Tryptophan. Die Spezifität der Beladung garantiert, daß während der Translation durch eine Codon-Anticodon-Erkennung die richtige, d.h. dem genetischen Code eines Gens entsprechende Aminosäure für die Proteinsynthese verwendet wird.
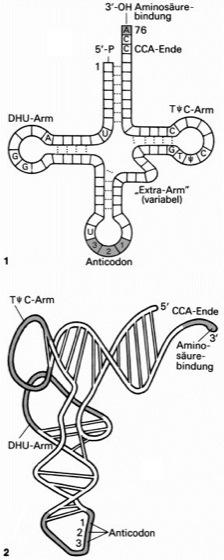
transfer-RNA
Abb. 2:
1Sekundärstruktur („Kleeblattstruktur“) von tRNA. Bei allen tRNA-Molekülen bildet das 3'-Ende die Sequenz 3'-ACC. Das 3'-terminale Adenin ist vom ersten Nucleosid der TψC-Schleife (Ribothymidin oder Uridin) immer 21 Nucleoside entfernt. Das 5'-Ende ist immer phosphoryliert. Ein Aminoacyl-tRNA-Molekül, das an das Ribosom bindet und aktiv an der Proteinbiosynthese teilnimmt, trägt die Aminosäure als Ester in Position 3' des 3'-terminalen Adenosinrests. Durch intramolekulare Basenpaarungen (Pünktelung) falten sich die tRNA-Ketten zur Kleeblattstruktur, wobei in Einzelfällen auch nicht-klassische Basenpaare (z.B. G–U-Paare) auftreten (Basenpaarung [Abb.]). Das Nucleotid am 5'-Ende trägt die Nummer 1; die Nummern 34-36 bilden das Anticodon, das zum entsprechenden Codon der mRNA komplementär ist. Die den beiden Enden benachbarten Bereiche vereinigen sich zu einem aus 7 Basenpaaren bestehenden „Stamm“ (im Bild oben), der wegen des angrenzenden 3'-Endes, an dem die jeweilige Aminosäure gebunden wird, als Akzeptor-Stamm (A-Stamm) bezeichnet wird. Die übrigen 3 durch Basenpaarung charakterisierten Bereiche („Blätter“ des Kleeblatts) werden als DHU-Arm (D-Arm, bestehend aus DHU-Schleife und -Stamm), Anticodon-Arm (AC-Arm, bestehend aus Anticodon-Schleife und -Stamm) und TψC-Arm (T-Arm, bestehend aus TψC-Schleife und -Stamm) bezeichnet. Der sog. Extra-Arm ist in seiner Länge variabel (bis zu 19 Nucleotide) und wird daher auch als V-Arm bezeichnet. Die Bezeichnungen DHU-Arm und TψC-Arm leiten sich von den in diesen Armen vorkommenden modifizierten Nucleosiden (Basenmodifikation, modifizierte Basen; DHU = Dihydrouridin, T = Ribothymidin, ψ = Pseudouridin) ab.
2Tertiärstruktur („L“-Form) von tRNA. Je 2 der gepaarten Bereiche der Kleeblattstruktur sind zu nahezu durchgehenden Helixstrukturen aufeinander „gesteckt“: der Akzeptor-Stamm und der TψC-Stamm bilden die aus insgesamt 12 Basenpaaren aufgebaute obere Querleiste, während der Anticodon-Stamm zusammen mit dem DHU-Stamm die im Bild senkrecht stehende Helix bilden. Die Gesamtstruktur ist durch zahlreiche, im Bild nicht gezeigte Wasserstoffbrückenbindungen stabilisiert. Der Abstand zwischen Anticodon und 3'-Ende beträgt etwa 8 nm.

transfer-RNA
Abb. 3: Lineare Nucleotidsequenz eines tRNA-Moleküls. Die Lage von DHU-Arm, Anticodon-Arm, TψC-Arm und dem CCA-Ende, an dem die Aminoacylierung erfolgt, sind von links nach rechts (5'-3'-Richtung) hervorgehoben.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.