Lexikon der Chemie: Nucleinsäuren
Nucleinsäuren, Biopolymere, bei denen Nucleoside über 3',5'-Phosphorsäure-diesterbrücken linear miteinander verbunden sind (Abb. 1). Nach dem an die Nucleosidbasen glycosidisch gebundenem Zucker werden Ribonucleinsäuren (Abk. RNS oder RNA, engl. ribonucleic acid) und Desoxyribonucleinsäuren (Abk. DNS oder DNA, engl. deoxyribonucleic acid) unterschieden. An die Polyester-Hauptkette der N. sind als Seitengruppen glycosidisch die Nucleinsäurebasen gebunden. Die typischen N. enthalten als Purinbasen Adenin und Guanin, als Pyrimidinbasen Cytosin sowie Uracil (nur in RNA) oder Thymin (nur in DNA); Tab. 1. RNA und DNA enthalten ferner noch Nebenbasen, insbesondere methylierte Derivate der Pyrimidine (in 3-Stellung sowie an der exocyclischen Aminogruppe) und Purine (in 1- und 7-Stellung sowie an der exocyclischen Aminogruppe), verschiedene in 5-Stellung substituierte Pyrimidine, Thiouracil und das C-Glycosid Pseudouridin. Die Methylierung der DNA scheint eine Rolle bei der Regulation der Transcription zu spielen. Besonders reich an Nebenbasen (bis zu 10 %) ist die tRNA. N. lassen sich säurekatalysiert hydrolysieren. Es wird zunächst bevorzugt die Phosphorsäureestergruppierung in 5'-Stellung gespalten, so daß primär die 3'-Phosphate entstehen. Die Hydrolyse verläuft weiter über die Nucleoside bis zu den freien Basen. DNA werden leichter als RNA hydrolysiert. N. reagieren sauer. Sie liegen in der Zelle gewöhnlich salzartig, insbesondere an basische Proteine gebunden, vor.
Nucleinsäurehydrolysate dienen zur Gewinnung von Nucleinsäurebasen, Nucleosiden und Nucleotiden. Zum Beispiel ergibt die Behandlung von N., meist werden die billigen Hefenucleinsäuren eingesetzt, mit Trichloressigsäure bei 0 °C eine säurelösliche Nucleotid-Fraktion, aus der die Adenosinphosphorsäuren isoliert werden können. Zur Isolierung von Nucleotiden aus Nucleinsäurehydrolysaten dienen Fällungsmethoden sowie die Ionenaustauschchromatographie. Durch Hydrolyse mit wäßrigem Ammoniak oder Pyridin bei erhöhter Temperatur und unter Druck werden die Nucleoside erhalten.
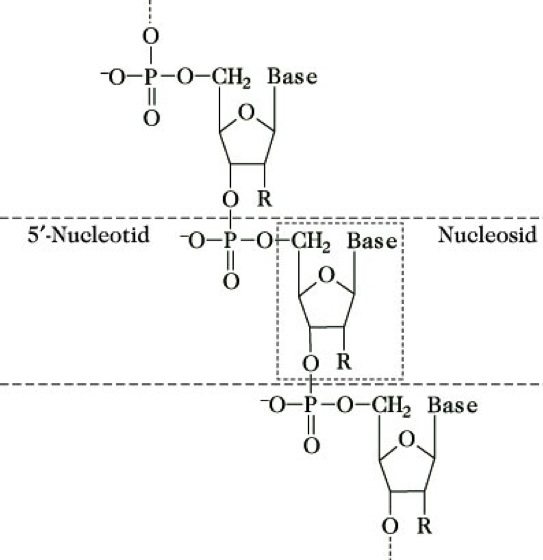
Nucleinsäuren. Abb. 1: Schematischer Aufbau einer Ribonucleinsäure (R = OH) und Desoxyribonucleinsäure (R = H).
Nucleinsäuren. Tab. 1: Bausteine der Nucleinsäuren.
| ||||
| Name | Abk. | Name | Abk. | Kurzform |
| Adenin | Ade | Adenosin | Ado | A |
| Guanin | Gua | Guanosin | Guo | G |
| Cytosin | Cyt | Cytidin | Cyd | C |
| Thymin | Thy | Ribosylthymin | Thd | T |
| Uracil | Ura | Uridin | Urd | U |
Desoxyribonucleinsäuren. Die Hauptmenge der DNA ist in den Chromosomen, also bei den Eukaryoten im Zellkern lokalisiert. Neben dieser chromosomalen DNA gibt es noch Satelliten-DNA, zu der die DNA der Chloroplasten (bei Pflanzen) oder Mitochondrien (mtDNA, bei Tieren) sowie die Plasmide der Bakterien gehören. Das aus dem Zellkern zunächst isolierbare Material wird als Chromatin bezeichnet. Es enthält außer DNA noch geringe Mengen RNA sowie vor allem Proteine: die stark basischen Histone sowie Nichthistonproteine. Diese Bestandteile liegen im Chromatin eng gepackt vor. Die Länge der gestreckten DNA-Doppelhelix würde etwa 2 m betragen. Das Watson-Crick-Modell sieht für die DNA eine Doppelhelix aus gegenläufigen
(antiparallelen) komplementären und rechtsgängig verlaufenden Strängen vor, die durch Wasserstoffbrücken zwischen den komplementären Basen
(A : T = 2, G : C = 3) zusammengehalten werden (Watson-Crick-Basenpaarungen) (Abb. 2). Das Verhältnis von [A + G]/[C + T] ist immer 1; [A + T]/ [C + G] schwankt im Bereich zwischen 0,3 und 2,5.
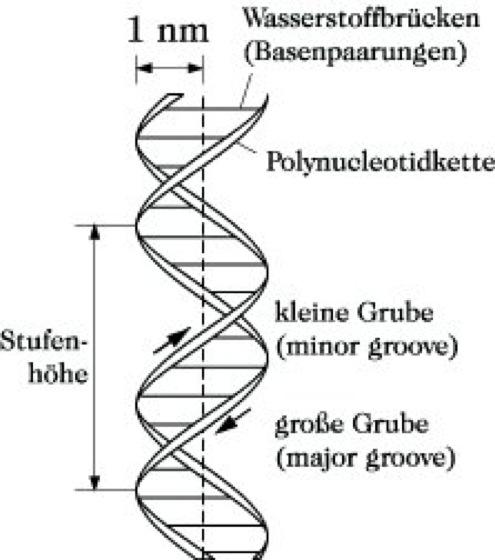
Nucleinsäuren. Abb. 2: DNA-Doppelhelix.
Die DNA-Doppelhelix (Abb. 2) kann in verschiedenen Formen (A, B, C, D, Z) vorliegen, deren Strukturparameter (Tab. 2) aus Röntgenfaserdiagrammen hervorgingen. Nativ liegt die DNA wahrscheinlich bevorzugt in der B-Form vor, die stark hydratisiert ist (90 % Wassergehalt als Na-Salz oder nach Li+-Zusatz). Bei der Präparation wandelt sich die B-Form in Abhängigkeit vom Wassergehalt und von der Elektrolytkonzentration in die C- oder A-Form um. Hybride aus komplementären DNA- und RNA-Strängen liegen unabhängig von der Ionenstärke in der A-Form vor. Im Unterschied zur A-, B-, C- und D-Form ist die später entdeckte Z-Form ("Z" für Zickzack-Kurs des Desoxyribosephosphat-Rückgrates) linksgängig. Sie wurde für Polynucleotide wie Poly(dG-dC)·Poly(dG-dC) oder Poly(dG-dT)·Poly-(dA-dC) gefunden (Rich). In der Z-Form liegen die Purinnucleoside in der syn-Konformation vor, die sich durch Drehung um die glycosidische Bindung von der sonst bei Purinen üblichen anti-Konformation unterscheidet. Der Übergang von der B- in die Z-Form scheint durch Methylierung der Basen (Bildung von Nebenbasen) begünstigt zu werden.
Nahezu das gesamte Erbgut aller Organismen ist in Form doppelsträngiger DNA gespeichert, die häufig, z. B. in Plasmiden, ringförmig geschlossen ist (ccc-Moleküle, von engl. covalently closed circles). Derartige Moleküle sind meist in sich noch weiter verflochten und bilden hinsichtlich der Drehrichtung einer rechtsgängigen Helix zusätzlich meist negativ überwundene Superhelices (engl. supercoils) aus. Die überspiralisierten Strukturen entstehen, wenn die Helix in einem linearen DNA-Molekül mehrere Male entwunden wird, bevor die Enden miteinander verknüpft werden. In vivo sind an diesem Prozeß mehrere Enzyme (DNA-Gyrasen und -Topoisomerasen) beteiligt.
An die DNA-Doppelhelix können aufgrund ihrer Struktur und spezifischen Wechselwirkungen mit den Basen oder/und dem Zuckerphosphat-Rückgrat eine Vielzahl von Proteinen (DNA-bindende Proteine) binden und so die Transcription regulieren.
Beim Erwärmen einer DNA-Lösung entwindet sich die Doppelhelixstruktur, die Basen-Paarung geht verloren und die DNA zerfällt in Einzelstränge ("Schmelzen der DNA"). Durch die Bildung von Einzelsträngen kommt es zu einem Anstieg der Lichtabsorption bei 260 nm (Hyperchromizität), so daß der Schmelzvorgang optisch verfolgt werden kann.
Nucleinsäuren. Tab. 2: Strukturparameter der verschiedenen Formen der DNA-Doppelhelix.
| ||||||
| Steighöhe der Helix (in nm) | 1,1 | 1,0 | 0,933 | 0,8 | 1,2 | |
| Anzahl der Basen je Steighöhe | 28,2 | 34 | 30,7 | 24 | 44,5 |
Ribonucleinsäuren. Die RNA kommen vor allem im Cytoplasma, vorwiegend in den Ribosomen, den Orten der Proteinsynthese, vor. Nach ihrer Funktion werden Boten- oder messenger RNA (Abk. mRNA), ribosomale RNA (Abk. rRNA) und transfer-RNA (Abk. tRNA) unterschieden. Die mRNA ist ein Abdruck der chromosomalen DNA und dient als Matrize für die Proteinsynthese. Die tRNA hat die Aufgabe, die für die Proteinsynthese benötigten Aminosäuren spezifisch zu binden (z. B. tRNAPhe → Phe-tRNAPhe), damit gleichzeitig zu aktivieren und durch die spezifische Codon-Anticodon-Wechselwirkung an die mRNA zu bringen. Die Bindung an die mRNA erfolgt dabei durch spezifische Basenpaarung des aminosäurespezifischen Basentripletts an das komplementäre Anticodon der tRNA. Geringe Mengen RNA finden sich ferner noch in den Mitochondrien sowie im Zellkern. Die Kern-RNA (Abk. nRNA) ist ein Vorläufer der mRNA. Als besondere Gruppe kann ferner noch die RNA der Viren und Viroide unterschieden werden. Im Unterschied zur DNA ist die RNA im allg. einsträngig. Diese kann innerhalb bestimmter Bereiche mit internen komplementären Sequenzen Doppelhelixstrukturen ausbilden (Basenpaarung G : C und A : U). Solche Strukturen sind für bestimmte Funktionen verantwortlich, z. B. die Haarnadelschleife (Abb. 3), die die Termination der RNA-Biosynthese durch die RNA-Polymerase auslöst sowie die Kleeblatt-Strukturen der tRNA. Doppelhelixstrukturen ergeben sich auch bei RNA-DNA-Hybriden, z. B. bei der DNA-abhängigen RNA-Synthese durch die RNA-Polymerasen. Letztere finden auch bei Hybridisierungsverfahren praktische Anwendung.
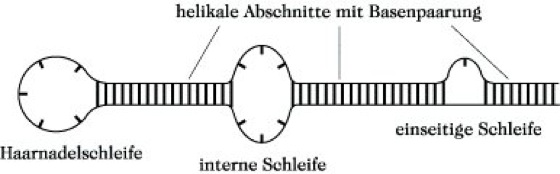
Nucleinsäuren. Abb. 3: Sekundärstrukturen von einsträngiger RNA.
Einige tierische und pflanzliche Viren enthalten doppelsträngige RNA, deren Doppelhelix der A-Form der DNA-Doppelhelix ähnelt. Die RNA sind globulär angeordnet. tRNA sind mit etwa 80 Nucleotiden und einer Mr von etwa 25000 relativ kleine Moleküle, deren Sekundärstruktur einem Kleeblatt entspricht. Etwa die Hälfte der Basen gehört helikalen Strukturen an. Die Nebenbasen sind in den Schleifen lokalisiert. Die Mr der mRNA beträgt etwa 106. Die Sekundärstruktur ist wesentlich komplizierter als die der DNA. In den rRNA liegen große Teile als Doppelhelix vor.
Sequenzanalysen sind für RNA und DNA möglich. Von besonderer Bedeutung sind DNA-Sequenzanalysen. Dafür werden zwei methodisch unterschiedliche Verfahren angewendet: 1) Bei der Maxam-Gilbert-Technik (chemische DNA-Sequenzierung) erfolgt die basenspezifische Spaltung der DNA mit Hilfe von organischen Reagenzien (G-spezifische Reaktion – Behandlung mit Dimethylsulfat; (G + A)-spezifische Reaktion – Behandlung mit Ameisensäure; (T + C)-spezifische Reaktion – Behandlung mit Hydrazin). 2) Beim Sanger-Verfahren (enzymatische DNA-Sequenzierung, Didesoxy-Sequenzierung, Kettenabbruch-Verfahren) werden zur Sequenzierung DNA-Polymerasen eingesetzt.
Biosynthese. Die DNA ist der Speicher der genetischen Information der Zelle (Genom). Eine Ausnahme bilden die RNA-Viren, bei denen aber vor der Genexpression, dem Ablesen der genetischen Information durch Nucleinsäure- und Proteinsynthese, erst in einer Wirtszelle die Synthese einer komplementären DNA aus dieser Viren-RNA in Anwesenheit der reversen Transcriptase erfolgt.
Die DNA-Synthese, die vor der Zellteilung zu einer Verdopplung der DNA führt, wird als Replikation, die RNA-Synthese wird als Transcription bezeichnet. Für Replikation und Transcription sind außer den Matrizen (DNA) noch die Bausteine, die entsprechenden Nucleosidtriphosphate, sowie Polymerasen erforderlich.
Am einfachsten erfolgt die RNA-Synthese bei den Prokaryoten (Bakterien, Blaualgen). Eine Informationseinheit der DNA wird als Operon bezeichnet. Produkte der Transcription sind die mRNA, die rRNA und die tRNA, die dann für die Proteinsynthese (Translation, Proteine) benötigt werden. Der eigentliche Informationsträger für die Proteinsynthese ist dabei die mRNA, deren Sequenz der des entsprechenden DNA-Abschnittes komplementär ist. Mit dem 5'-Ende wird die mRNA an das Ribosom als den Ort der Proteinsynthese gebunden.
Bei den Eukaryoten verläuft die Nucleinsäuresynthese komplizierter. Hier sind neben DNA-Abschnitten, die als Sequenzinformation im Protein auftauchen (Exons), noch Abschnitte enthalten, die nicht zur Synthese des Proteins abgelesen werden (Introns). Ort der Replikation und Transcription ist der Zellkern.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.