Molekularbiologie: Revolution in der Proteinforschung
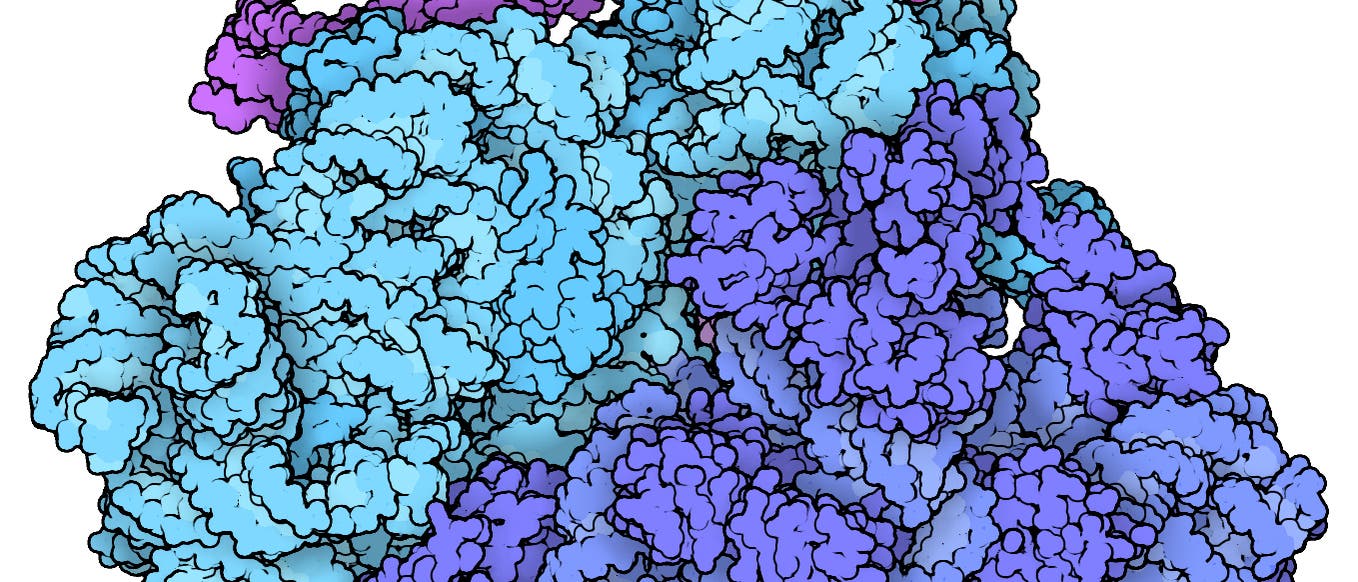
Seit mehr als zehn Jahren versuchen der Molekularbiologe Martin Beck und seine Kollegen, eines der schwierigsten Puzzles der Welt zusammenzusetzen: ein detailliertes Modell vom Kernporenkomplex, einer riesigen Kanalstruktur, die zu den größten molekularen Maschinen in menschlichen Zellen zählt. Dieser gigantische Apparat steuert, welche Moleküle in den Zellkern eintreten beziehungsweise ihn verlassen. In jeder Zelle gibt es hunderte solcher Komplexe. Sie bestehen aus je rund tausend Proteinmolekülen, die sich zu einer Struktur ähnlich einem Hohlzylinder zusammenfügen, der die innere mit der äußeren Kernmembran verbindet und einen Durchgang durch die Kernhülle erzeugt.
Mehr als 30 Sorten von Eiweißen, von denen jeweils mehrere Kopien vorliegen, bilden einen Kernporenkomplex. Sie werden als Nukleoporine (abgekürzt Nup) bezeichnet und sind vielfältig miteinander verbunden. Ihre räumlichen Formen sind zwar experimentell ermittelt worden, allerdings anhand von Proben verschiedener biologischer Arten, weshalb nicht klar ist, wie sie innerhalb ein und desselben Organismus zusammenwirken. Man weiß, wie das fertig zusammengesetzte Puzzle in etwa aussieht, da es niedrig aufgelöste 3-D-Darstellungen vom Kernporenkomplex gibt. Doch die sind nicht detailreich genug, um zu erkennen, wie sich die Teile im Einzelnen zusammenfügen.
Im Jahr 2016 stellte Beck, der am Max-Planck-Institut für Biophysik (MPIBP) in Frankfurt arbeitet, gemeinsam mit seinem Team ein räumliches Modell des Kernporenkomplexes vor. Es deckte etwa ein Drittel der Struktur dieses molekularen Apparats und rund die Hälfte der Nup-Proteine ab. Das war damals ein bedeutender Fortschritt …
Schreiben Sie uns!
Beitrag schreiben