3-D-Druck: Forscher bauen Minizoo für Bakterien
Wenn sie in ihrem natürlichen Element sind - das heißt zum Beispiel im menschlichen Körper -, verhalten sich Bakterien oft ganz anders als in der Petrischale. Der Grund sind die Mikrostrukturen, die Nischen, die sie dort besiedeln und in denen sie sich mit anderen Erregern zusammentun. Damit sich diese Vorgänge besser im Labor untersuchen lassen, haben Wissenschaftler um Jason Shear von der University of Texas in Austin nun ein Verfahren zum Bau winziger 3-D-Strukturen entwickelt. Sie sollen die Lebensbedingungen von Bakterien simulieren.
Zum Bau ihrer einige zehn Mikrometer großen Strukturen lassen die Forscher den gewünschten Erregerstamm zunächst in einer erwärmten und daher flüssigen Gelatinemischung wachsen. Durch Abkühlung erhalten sie einen festen Block, in den sie mit einem Laser wie bei einem 3-D-Drucker die Struktur einschreiben: Wo der Lichtstrahl fokussiert wird, bilden lichtempfindliche Moleküle in der Gelatine eine netzartige Struktur aus und stabilisieren sie. Die Bakterienkäfige können dadurch eine nahezu beliebige Form annehmen.
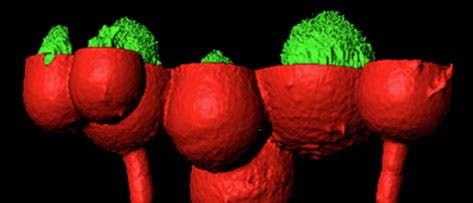
Die Mikroskopaufnahmen zeigen diverse Gehäuseformen (rot), in denen die Forscher Kolonien von P. aeruginosa (grün) heranzüchteten. Jede Kapsel hat ein Volumen von wenigen Pikolitern, der Maßstabsbalken entspricht 20 Mikrometern.
Im Bild unten rechts ist erkennbar, wie Bakterien aus miteinander verbundenen Kompartimenten zu Artgenossen wandern, um eine größere Kolonie zu bilden.
Durch erneutes Erwärmen werden schließlich die nicht verfestigten Gelatinestellen wieder verflüssigt und mit einem Nährmedium ausgewaschen. Zurück bleibt die räumliche Struktur, die der Laser abgefahren hatte. Die eingeschlossenen Bakterien können sich darin frei bewegen und reifen abschließend zu einer "Mikrokolonie" heran.
Ein Vorteil des Materials sei, dass es zwar die Bakterien an der Ausbreitung hindere, nicht aber die Diffusion von Nähr-, Abfall- und Signalstoffen unterbinde. So kann die Reaktion der Kolonien auf äußere Einflüsse studiert werden. Außerdem ist es möglich, unterschiedliche Arten von Erregern in benachbarten Abschnitten einzuschließen. Es entstünden komplette mikrobielle Ökosysteme, schreiben die Forscher.
Dies demonstrierten sie durch Nachbau einer typischen Form des Bakterienbefalls im menschlichen Körper und in medizinischen Geräten: eine Koinfektion von Staphylococcus aureus und Pseudomonas aeruginosa. Sie lässt sich mit bestimmten Antibiotika nur schwer bekämpfen, weil resistente P. aeruginosa die eingeschlossenen Staphylokokken mitschützen. Diesen Effekt konnte das Team um Shear reproduzieren, indem sie eine Staphylokokken-Kolonie in eine Schale aus P. aeruginosa gaben. Es zeigte sich, dass das Antibiotikum (Ampicillin) auf seinem Weg in den Kern der Struktur von den Pseudomonas-Bakterien, ganz wie erwartet, unschädlich gemacht wurde.
Für andere Experimente können im Gelatineblock besonders interessante Zellen erst identifiziert und dann mit Hilfe des 3-D-Drucks isoliert werden. In den Mikrokolonien wachsen dadurch ausschließlich Nachkommen dieses einen Bakteriums. Auch durch kleine Kanälchen verbundene "Habitate" sind möglich. Weil es vom Design der Struktur bis zum Druck nur wenige Minuten dauere, könne man recht flexibel ausprobieren, welche Form sich im jeweiligen Fall am besten eigne.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.