Zellphysiologie: Zoom in die Zellarbeit
Erst sehen heißt wissen, findet so mancher Enthüllungsreporter zellulärer Geheimnisse. Und genauer hinkucken zu können, heißt demnach auch mehr Chancen auf wirklich exklusive Sensationen. Da kommt ein neues Teleobjektiv kommt allen Zellinterna-Papparazzi sehr gelegen.
© Block et al. (Ausschnitt)
Wie aus Informationen Gene werden und umgekehrt oder wie aus DNA Proteine werden und warum – darüber sind ja nun wirklich schon Bände geschrieben worden, die ein paar Bibliotheken füllen. Details finden sich in allen Kapiteln genug – und zwar derart viele, dass leicht übersehen wird, wie viele Wissenslücken zwischen den Zeilen lauern.
So ist zum Beispiel die so genannte Transkription eigentlich ein alter Hut – bei dem Vorgang wird die unbewegliche DNA mit ihren Informationen kopiert in die bewegliche Bauanleitung RNA, die dann zu den Arbeitern der Eiweißfabrikationshallen vor den Toren des Zellkerns geschickt wird. Zuständig für die Transkription ist ein Zellarbeiter namens RNA-Polymerase.
Wie dieses Enzym arbeitet, ist extrem detailliert untersucht: Es sorgt dafür, dass aus einem Buchstaben des stabilen Informationsträgers DNA ein entsprechendes Gegenstück des mobilen Informationsüberträger RNA geblaupaust wird. Zu diesem Zweck hangelt sich die RNA-Polymerase ablesend an der DNA entlang und spuckt am anderen Ende einen Lochstreifen RNA aus, der dann nach ein paar Schönheitskorrekturen zu den ribosomalen Proteinproduzenten der Zelle gelangt. Das alles könnten Sie auch von aufmerksamen Biologieschüler erfahren, oder in einem der erwähnten Bände in einer der vielen Bibliotheken en detail nachlesen. Also zum Spannenden, den Lücken zwischen den Details.
Zum Beispiel dieser: Fragt man Schüler, Lehrer oder Professoren der Zellbiologie einfach danach, wie die RNA-Polymerase sich eigentlich vorwärts bewegt, dann müssen alle mit den Achseln zucken. Irgendwie kommt sie halt voran, ruckelnd, gleitend oder sonst wie.
Dabei ist schon bemerkenswert genug, dass sie dies überhaupt hinbekommt – hierfür sollte irgendeine Art von Energieverbrauch nötig sein, die aber nach allen bisherigen Erkenntnissen der RNA-Polymerase durch keine bekannten zellulären Brennstoffe zur Verfügung gestellt wird. Wie kann das funktionieren?
Um erst einmal fundierte Hypothesen der RNA-Polymerasebewegung entwickeln zu können, wollten zellbiologische Theoretiker sich daher schon seit Längerem erst einmal am arbeitenden Objekt ansehen, ob das Enzym sich sprunghaft über ein paar gleichzeitig verarbeitete Basen oder eher gleichmäßig-gleitend Sprosse für Sprosse am DNA-Strang entlang bewegt – und scheiterten allein schon an den winzigen Dimensionen des Problemfalls.
Zur Verdeutlichung: Eine Streckenlänge von DNA-Base zu DNA-Base beträgt gerade einmal 0,34 Nanometer; selbst mit den besten, hochauflösensten aller optisch abbildenden Methoden muss aber die knapp dreifache Strecke zurückgelegt sein, bevor überhaupt ein Bewegungsschritt wahrgenommen werden kann. Der Effekt ist etwa der einer Videowiedergabe auf einem schmalbrüstigen Computer – hin und wieder ruckelt ein Bild hinein, alles dazwischen Liegende ist nicht zu erkennen. Hochauflösendere Verfahren taten dringend not.
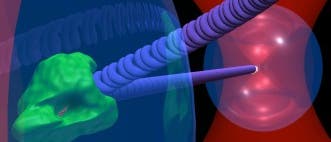
Abhilfe schufen nun Steven Block und seine Kollegen von der Stanford-Universität [1]. Sie beschäftigten sich bereits seit einiger Zeit mit so genannten optischen Fallen zur Abbildung extrem winziger Details – und verbesserten nun die Auflösung mit ein paar technischen Kniffen um den Faktor zehn.
Bei einer optischen Falle werden mit Hilfe der vermeintlich geringen Kraft eines Lichtstrahls Objekte im Nanomaßstab lange genug fixiert, um sie mit einem Mikroskop aufs Atom genau darstellen zu können – aus einem verwaschenen Schatten schält sich Detailreichtum. Die Technik stößt allerdings auf physikalische Grenzen. So flackert etwa in normaler Luft schon der eingesetzte Laserstrahl der Lichtpinzetten untolerierbar – Block und Kollegen packten den optischen Aufbau daher in ein Milieu reinen Stickstoffs, der den Lichtstrahl deutlich weniger stark streute. Mit weiteren Kniffen erreichten die Forscher nun letztlich eine Auflösung von rund 0,1 Nanometern – was etwa dem Durchmesser eines einzelnen Wasserstoffatoms entspricht.
Zum ersten Test-Ernstfall für das neue optische Werkzeug kürten die Wissenschaftler dann eben eine arbeitende, bakterielle RNA-Polymerase, die auf einem optisch fest getackerten DNA-Strang entlang zu eilen versucht – und schafften es, die Reise des Enzyms auf der DNA-Schiene ruckelfrei zu bewundern [2]. Klar wurde schnell eines: Einige der bis dato hoch gehandelten Erklärungsversuche der RNA-Polymerase-Lokomotion sind schlicht falsch. Die RNA-Polymerase bewegt sich etwa nicht wie unser Auge beim Lesen einer Buchzeile in Sprüngen, sondern vielmehr stets in gleichmäßigen 0,34-Nanometer-Schritten vorwärts. Sie arbeitet also immer fein säuberlich eine Basensprosse der DNA-Leiter nach der nächsten ab.
Aber hilft das nun bei der Erklärung, woher die RNA-Polymerase überhaupt den Brennstoff für ihr Fortkommen bezieht? Die Forscher bieten eine verblüffend schlichte Erklärung an: Wärme bringt das Enzym auf Trapp, genauer die Brown'sche Molekularbewegung. Die ist natürlich ungerichtet – zufällige Wärmestöße schubsen dabei ein jedes Objekt, das klein genug ist, mal hierhin, mal dorthin. Wie daraus dennoch eine gerichtete Bewegung entstehen kann, beschreiben die Forscher mit einem hüpfenden Gummiball, der zwischen zwei parallelen Mauern hin und her prallt: Bewegte man die linke Mauer jedes Mal ein Stück nach rechts, sobald der Ball davon abgeprallt ist (und zugleich auch die rechte), dann würde der Ball in der Summe insgesamt immer mehr nach rechts wandern, solange er überhaupt in Bewegung bleibt – dafür aber sorgt die omnipräsent in alle Richtungen schubsende Wärme.
Vielleicht fungiert eine DNA-Base, die von der anrückenden RNA-Polymerase erreicht wird, als eine Art Anker, der das Enzym nur nach einem Brown'schen Molekularschubs in eine einzige Richtung weiterziehen lässt: zur nächsten Base, also dem nächsten Anker. Zur Bewegung selbst müsste dann gar kein RNA-Polymerase-Benzin in Form von zellulären Energiemolekülen zugesetzt werden, glauben die Wissenschaftler. Ein wärmegespeistes Perpetuum mobile ist das ganze System dennoch nicht: Energie müsste die Zelle schließlich weiterhin für andere Prozesse aufwenden – etwa das Entwirren der verpackten DNA, dem Abbremsen oder vielleicht dem Aufspringen auf die abzulesende Matrize. An derartige Fragen, so hoffen die Wissenschaftler, könnte man sich ja vielleicht bei nächsten Mal mit der neuen Methode heranzoomen.
So ist zum Beispiel die so genannte Transkription eigentlich ein alter Hut – bei dem Vorgang wird die unbewegliche DNA mit ihren Informationen kopiert in die bewegliche Bauanleitung RNA, die dann zu den Arbeitern der Eiweißfabrikationshallen vor den Toren des Zellkerns geschickt wird. Zuständig für die Transkription ist ein Zellarbeiter namens RNA-Polymerase.
Wie dieses Enzym arbeitet, ist extrem detailliert untersucht: Es sorgt dafür, dass aus einem Buchstaben des stabilen Informationsträgers DNA ein entsprechendes Gegenstück des mobilen Informationsüberträger RNA geblaupaust wird. Zu diesem Zweck hangelt sich die RNA-Polymerase ablesend an der DNA entlang und spuckt am anderen Ende einen Lochstreifen RNA aus, der dann nach ein paar Schönheitskorrekturen zu den ribosomalen Proteinproduzenten der Zelle gelangt. Das alles könnten Sie auch von aufmerksamen Biologieschüler erfahren, oder in einem der erwähnten Bände in einer der vielen Bibliotheken en detail nachlesen. Also zum Spannenden, den Lücken zwischen den Details.
Zum Beispiel dieser: Fragt man Schüler, Lehrer oder Professoren der Zellbiologie einfach danach, wie die RNA-Polymerase sich eigentlich vorwärts bewegt, dann müssen alle mit den Achseln zucken. Irgendwie kommt sie halt voran, ruckelnd, gleitend oder sonst wie.
Dabei ist schon bemerkenswert genug, dass sie dies überhaupt hinbekommt – hierfür sollte irgendeine Art von Energieverbrauch nötig sein, die aber nach allen bisherigen Erkenntnissen der RNA-Polymerase durch keine bekannten zellulären Brennstoffe zur Verfügung gestellt wird. Wie kann das funktionieren?
Um erst einmal fundierte Hypothesen der RNA-Polymerasebewegung entwickeln zu können, wollten zellbiologische Theoretiker sich daher schon seit Längerem erst einmal am arbeitenden Objekt ansehen, ob das Enzym sich sprunghaft über ein paar gleichzeitig verarbeitete Basen oder eher gleichmäßig-gleitend Sprosse für Sprosse am DNA-Strang entlang bewegt – und scheiterten allein schon an den winzigen Dimensionen des Problemfalls.
Zur Verdeutlichung: Eine Streckenlänge von DNA-Base zu DNA-Base beträgt gerade einmal 0,34 Nanometer; selbst mit den besten, hochauflösensten aller optisch abbildenden Methoden muss aber die knapp dreifache Strecke zurückgelegt sein, bevor überhaupt ein Bewegungsschritt wahrgenommen werden kann. Der Effekt ist etwa der einer Videowiedergabe auf einem schmalbrüstigen Computer – hin und wieder ruckelt ein Bild hinein, alles dazwischen Liegende ist nicht zu erkennen. Hochauflösendere Verfahren taten dringend not.
Abhilfe schufen nun Steven Block und seine Kollegen von der Stanford-Universität [1]. Sie beschäftigten sich bereits seit einiger Zeit mit so genannten optischen Fallen zur Abbildung extrem winziger Details – und verbesserten nun die Auflösung mit ein paar technischen Kniffen um den Faktor zehn.
Bei einer optischen Falle werden mit Hilfe der vermeintlich geringen Kraft eines Lichtstrahls Objekte im Nanomaßstab lange genug fixiert, um sie mit einem Mikroskop aufs Atom genau darstellen zu können – aus einem verwaschenen Schatten schält sich Detailreichtum. Die Technik stößt allerdings auf physikalische Grenzen. So flackert etwa in normaler Luft schon der eingesetzte Laserstrahl der Lichtpinzetten untolerierbar – Block und Kollegen packten den optischen Aufbau daher in ein Milieu reinen Stickstoffs, der den Lichtstrahl deutlich weniger stark streute. Mit weiteren Kniffen erreichten die Forscher nun letztlich eine Auflösung von rund 0,1 Nanometern – was etwa dem Durchmesser eines einzelnen Wasserstoffatoms entspricht.
Zum ersten Test-Ernstfall für das neue optische Werkzeug kürten die Wissenschaftler dann eben eine arbeitende, bakterielle RNA-Polymerase, die auf einem optisch fest getackerten DNA-Strang entlang zu eilen versucht – und schafften es, die Reise des Enzyms auf der DNA-Schiene ruckelfrei zu bewundern [2]. Klar wurde schnell eines: Einige der bis dato hoch gehandelten Erklärungsversuche der RNA-Polymerase-Lokomotion sind schlicht falsch. Die RNA-Polymerase bewegt sich etwa nicht wie unser Auge beim Lesen einer Buchzeile in Sprüngen, sondern vielmehr stets in gleichmäßigen 0,34-Nanometer-Schritten vorwärts. Sie arbeitet also immer fein säuberlich eine Basensprosse der DNA-Leiter nach der nächsten ab.
Aber hilft das nun bei der Erklärung, woher die RNA-Polymerase überhaupt den Brennstoff für ihr Fortkommen bezieht? Die Forscher bieten eine verblüffend schlichte Erklärung an: Wärme bringt das Enzym auf Trapp, genauer die Brown'sche Molekularbewegung. Die ist natürlich ungerichtet – zufällige Wärmestöße schubsen dabei ein jedes Objekt, das klein genug ist, mal hierhin, mal dorthin. Wie daraus dennoch eine gerichtete Bewegung entstehen kann, beschreiben die Forscher mit einem hüpfenden Gummiball, der zwischen zwei parallelen Mauern hin und her prallt: Bewegte man die linke Mauer jedes Mal ein Stück nach rechts, sobald der Ball davon abgeprallt ist (und zugleich auch die rechte), dann würde der Ball in der Summe insgesamt immer mehr nach rechts wandern, solange er überhaupt in Bewegung bleibt – dafür aber sorgt die omnipräsent in alle Richtungen schubsende Wärme.
Vielleicht fungiert eine DNA-Base, die von der anrückenden RNA-Polymerase erreicht wird, als eine Art Anker, der das Enzym nur nach einem Brown'schen Molekularschubs in eine einzige Richtung weiterziehen lässt: zur nächsten Base, also dem nächsten Anker. Zur Bewegung selbst müsste dann gar kein RNA-Polymerase-Benzin in Form von zellulären Energiemolekülen zugesetzt werden, glauben die Wissenschaftler. Ein wärmegespeistes Perpetuum mobile ist das ganze System dennoch nicht: Energie müsste die Zelle schließlich weiterhin für andere Prozesse aufwenden – etwa das Entwirren der verpackten DNA, dem Abbremsen oder vielleicht dem Aufspringen auf die abzulesende Matrize. An derartige Fragen, so hoffen die Wissenschaftler, könnte man sich ja vielleicht bei nächsten Mal mit der neuen Methode heranzoomen.

Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.