Lexikon der Biologie: blotting-Techniken
blotting-Techniken, Sammelbezeichnung für eine Reihe methodisch verwandter Techniken ( vgl. Tab. ), die häufig zur Untersuchung und Charakterisierung von biogenen Makromolekülen (Biopolymere; Desoxyribonucleinsäuren [DNA], Ribonucleinsäuren [RNA], Proteine) eingesetzt werden. Die blotting-Techniken beruhen im wesentlichen auf einer von E.M. Southern 1975 entwickelten Methodik für den Nachweis und die Analyse spezifischer DNA-Fragmente in genomischen oder anderen DNA-Präparationen (sog. Southern-Technik). Die hierbei eingesetzten Prinzipien werden in modifizierter Weise auch für Untersuchungen von RNA (Northern-Technik) und Proteinen (Western-Technik), aber auch zur Analyse von Protein-DNA-Interaktionen(South-Western-Technik) oder Protein-RNA-Interaktionen (North-Western-Technik;RNA-bindende Proteine) herangezogen (s. u.). Die Bezeichnungen für diese von der Southern-Technik abgeleiteten Verfahren entstanden unter Bezugnahme auf den Namen "Southern" in Anlehnung an die Himmelsrichtungen.
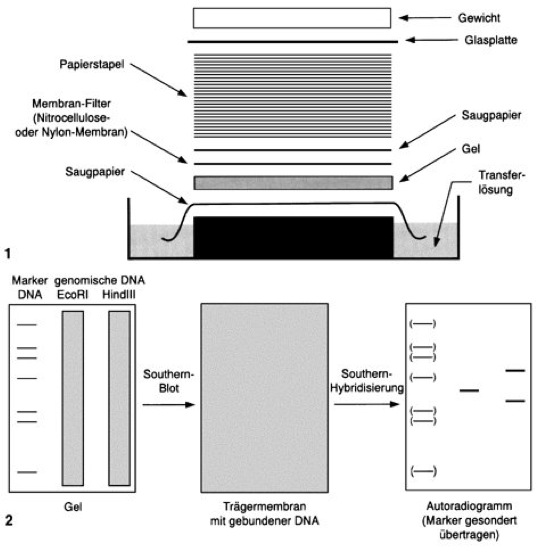
Die grundlegenden Schritte aller blotting-Techniken (Auftrennung, Transfer, Nachweis, vgl. Tab. ) sollen am Beispiel der Southern-Technik im Detail dargestellt werden: Die Southern-Technik zur Analyse von DNA kann in 3 wesentliche Teilschritte untergliedert werden: 1) Spaltung der zu untersuchenden DNA durch Restriktionsenzyme und Auftrennung der Fragmente durch Gelelektrophorese; 2) positionsgenauer Transfer der DNA-Fragmente auf eine geeignete Trägermembran durch "blotting" (s. u.) und Immobilisierung der DNA; 3) Nachweis von spezifischen Fragmenten in der gesamten Fragment-Population durch Hybridisierung mit markierten (Markierung) Sonden (Gensonde; Hybridisierungsprobe).
Zu 1): Die zu untersuchende DNA wird zunächst unter Verwendung von Restriktionsendonucleasen (Restriktionsenzymen) zu einem Fragmentgemisch gespalten. Dabei werden vollständige Spaltungen z. B. immer dann angestrebt, wenn unter Einsatz der Southern-Technik die Kopienzahl von Genen in einem Genom bestimmt werden soll. Unvollständige Spaltungen der eingesetzten DNA (partielle Spaltung) hingegen werden z. B. bei Restriktionskartierungen (Restriktionsanalyse) großer DNA-Bereiche durchgeführt. Das erhaltene Fragmentgemisch wird dann durch Gelelektrophorese (meist in Agarose-Gelen [Agar]; das hierbei benutzte Gel wird dann auch als "Southern-Gel" bezeichnet) zusammen mit Marker-DNA-Fragmenten bekannter Längen seiner Größe nach aufgetrennt. Bei Einsatz genomischer DNA (das Verfahren wird dann genomischer Southern genannt) ergibt sich aufgrund der immensen Vielzahl der entstehenden, unterschiedlich großen Fragmente meist eine mehr oder weniger einheitliche Bahn eng aneinandergereihter DNA-Bruchstücke, die optisch nicht mehr voneinander zu unterscheiden sind. Bisweilen in diesem "Schmier" hervortretende Banden lassen sich auf repetitive DNA oder Genfamilien zurückführen. Die gesuchten spezifischen Banden innerhalb dieses Schmiers werden durch die sich anschließenden Schritte erkennbar gemacht.
Zu 2): Der Transfer der aufgetrennten Fragmente auf geeignete Trägermembranen (wie Filter aus Nitrocellulose oder Nylon-Filter) unter Beibehaltung des Fragmentmusters kann als zentraler Schritt der Southern-Technik sowie der daran angelehnten Techniken angesehen werden und wird hier auch als Southern-Transfer oder Southern-Blot (Southern-Blotting) bezeichnet. Die ursprüngliche Methode für die positionsgenaue Übertragung des Fragmentmusters aus dem Gel auf eine Membran bedient sich des Kapillartransfers unter Verwendung von Nitrocellulose-Filtern als Trägermembran, des SSC-Puffers (SSC) als Transferlösung in hoch konzentrierter Form (z. B. 20 × SSC [3 M NaCl, 300 mM Trinatriumcitrat], da DNA bei geringer Ionenstärke schlechter an die Nitrocellulose bindet) und stark Flüssigkeit saugender Spezialpapiere. Die Effizienz der Übertragung insbesondere von sehr großen DNA-Fragmenten kann vor dem Transfer durch limitierte Einwirkung von Salzsäure und anschließende Alkali-Behandlung der DNA in dem Trenngel gesteigert werden. Durch die saure Hydrolyse entstehen zunächst einige depurinierte Basenpositionen, an denen dann das Zucker-Phosphat-Rückgrat der DNA gespalten wird; die so entstehenden kleineren Bruchstücke besitzen eine bessere Mobilität aus dem Gel auf die Membran, so daß der Transfer mit größerer Effizienz erfolgen kann. Außerdem wird durch diese Schritte, aber auch durch die Verwendung der sehr salzhaltigen Transferlösung sichergestellt, daß die DNA schließlich in denaturierter (Denaturierung), einzelsträngiger Form vorliegt. Dies wiederum ist wichtig für die spätere Hybridisierung mit der Sonde zum Nachweis spezifischer Fragmente. Bei der Kapillartransfer-Methode werden die DNA-Bruchstücke zum Transfer aus dem Gel in Richtung der Membran gesaugt ("geblottet", von englisch to blot = saugen; dieser "Saugtransfer" ist namengebend für die blotting-Techniken), indem durch den besonderen Aufbau verschiedener Komponenten ( vgl. Abb. ) bewirkt wird, daß in dem Fluß des Transferpuffers die DNA-Fragmente mitgeführt und an dem für sie undurchlässigen Membranfilter zurückgehalten und gebunden werden. Nach dem Zerlegen der Transferanordnung kann die Membran mit den daran haftenden, denaturierten DNA-Molekülen entnommen werden. Die Haftung der DNA an die Membran kann bei Verwendung von Nitrocellulose-Filtern durch "backen" (d. h. Erhitzen der Filter auf 60–70 °C unter Vakuum) weiter verstärkt werden. Bei den meisten Kunststoffmembranen (vor allem Nylon-Membranen) kann durch Bestrahlung mit UV-Licht (Wellenlänge meist 254 nm; UV-crosslinking; Cross-link) eine kovalente Vernetzung der DNA mit dem Membranfilter und entsprechend starke Bindung an die Membran erreicht werden. Die so auf der Trägerfolie immobilisierte und denaturierte DNA kann nun direkt für den nachfolgenden Nachweis durch Hybridisierung eingesetzt werden.
Zu 3): Der Nachweis spezifischer DNA-Moleküle auf dem Membranfilter bedient sich der Technik der molekularen Hybridisierung einer genau bekannten, markierten Gensonde an die zu ihr komplementären Zielsequenzen. Dieser Schritt wird hier auch als Southern-Hybridisierung bezeichnet. Als Sonden werden z. B. radioaktiv markierte DNA-Fragmente eingesetzt. In neuerer Zeit wird jedoch nach Möglichkeit mit nicht-radioaktiven markierten Sonden gearbeitet (Markierung). Bei der Hybridisierung werden die in Schritt 2) hergestellten Membranen unter je nach Sonde und Fragestellung unterschiedlichen Bedingungen ("Hybridisierungsbedingungen", im wesentlichen bestimmt durch: den G/C-Gehalt der Sonde, die Hybridisierungstemperatur, die Ionenkonzentration der Hybridisierungslösung, den Anteil an denaturierenden Substanzen wie z. B. Formamid in der Hybridisierungslösung; Stringenz) mit markierter Hybridisierungssonde (im engl. meist als "probe" bezeichnet) inkubiert. Um unspezifische Bindungen der Sonde an den Filter möglichst zu unterbinden und dadurch klare, spezifische Signale zu erhalten, werden der Hybridisierungslösung meist noch Blockierungsreagenzien, wie unspezifische DNA (z. B. Kalbsthymus-DNA) und Rinderserumalbumin ("BSA") oder Denhardts Lösung, zugegeben (blocking). Durch Basenpaarung der Sonde an ihre Zielsequenzen aufgrund der Komplementaritätsregel kann die Position der gesuchten, passenden DNA-Moleküle aus der gesamten Fragmentpopulation durch jeweils geeigneten Nachweis der Markierung der Sonde bestimmt werden. Durch Vergleich mit den Positionen bekannter Marker-Fragmente ist die Größe der gefundenen DNA-Moleküle zu ermitteln. Es können jedoch diese DNA-Fragmente auch aus der Trägermembran oder einem parallel bearbeiteten, identischen zweiten "Southern-Gel" für weiterführende Arbeiten isoliert werden. –
Die durch die Southern-Technik eingeführte, prinzipielle Methodik ist in verschiedene Richtungen abgewandelt worden und so nun in modifizierter Weise auch für die Handhabung und Analyse von weiteren Makromolekülen einsetzbar.
Bei der Northern-Technik handelt es sich um ein Verfahren zum Nachweis von RNA-Molekülen (Ribonucleinsäuren), also Transkripten eines Gens, unter Einsatz einer bekannten DNA, z. B. dem Gen selbst oder Teilen davon als Sonde. Die Bezeichnung "Northern" entstammt einem Wortspiel unter Bezugnahme auf "Southern" und deutet zugleich die Komplementarität der bei diesen beiden Techniken analysierten Moleküle an. Bei der Northern-Technik wird die aus Zellen oder Geweben isolierte RNA (anstelle von DNA) als Ausgangsmaterial und zu analysierendes Makromolekül eingesetzt. Die RNA wird auch hier zunächst gelelektrophoretisch aufgetrennt. Dabei wird die Gelelektrophorese jedoch unter denaturierenden Bedingungen, d. h. in Alkali-, Formaldehyd-, Glyoxal- oder Harnstoff-Gelen, durchgeführt. Hierdurch ist während des Gellaufs sichergestellt, daß RNA-Sekundärstrukturen, die das Laufverhalten der RNA-Moleküle verändern und falsche relative Molekülmassen vortäuschen, nicht auftreten können. Die aufgetrennten RNA-Moleküle werden dann durch der Southern-Technik entnommene Verfahren auf geeignete Membranen (wie bei der Southern-Technik Nitrocellulose- oder Nylon-Filter) geblottet und dort fixiert. Durch Inkubation der Membranen mit markierten DNA-Sonden im Rahmen der Hybridisierung können die entstandenen DNA-RNA-Hybride lokalisiert und die Transkripte des der Sonde zugrundeliegenden Gens nachgewiesen werden. Der Einsatz von ribosomalen RNAs oder anderen RNAs definierter Länge als Molekülmassen-Standards während der Gelelektrophorese erlaubt es, die detektierten RNA-Moleküle bestimmten Größen zuzuordnen. Ergänzend zu diesen qualitativen können auch quantitative Aussagen, z. B. über die Transkriptmenge und somit über die Aktivität des untersuchten Gens, durch Northern-Analysen erhalten werden. Zur weitergehenden Charakterisierung der RNA können z. B. S1-Kartierung und primer-extension-Analysen durchgeführt werden.
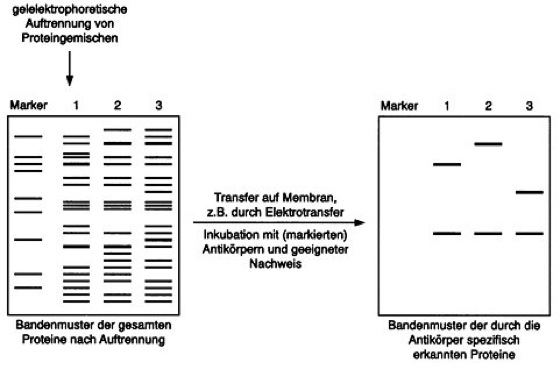
Durch die ebenfalls methodisch von der Southern-Technik (s. o.) abgeleitete Western-Technik kann eine dritte große Klasse biogener Makromoleküle, die Proteine, untersucht werden. Durch die Western-Technik ist es möglich, Proteingemische (z. B. Gesamtproteinextrakt nach einem Zellaufschluß) oder auch Einzelproteine qualitativ und/oder quantitativ zu analysieren. Auch hier wird zunächst das zu untersuchende Molekülgemisch durch eine Gelelektrophorese, meist durch eine denaturierende SDS-Polyacrylamid-Gelelektrophorese, aufgetrennt. Die aufgetrennten Proteine werden dann aus dem Gel auf eine geeignete Membran mit hohen Proteinbindungskapazitäten (oft Membranen aus PVDF) positionsgenau übertragen ("geblottet") und immobilisiert. Bei der Western-Technik erfolgt das blotting meist durch die Elektrotransfer-Methode ( vgl. Infobox ). Die so erhaltene Membran, der Western-Blot, kann nun für den Nachweis, die Lokalisation (und damit Bestimmung der Molekülmasse durch Vergleich der Positionen mit aufgetrennten Markerproteinen) und eventuell Quantifizierung bestimmter Proteine mit jeweils geeigneten Methoden eingesetzt werden. Bei einigen dieser nachfolgenden Analysen ist eine Vorbehandlung des Western-Blots zur Absättigung (Blockierung) unspezifischer Bindungsstellen auf der Membran analog der Inkubation einer bei der Southern-Technik hergestellten Membran vor den Nachweisreaktionen erforderlich. Als Blockierungsreagenzien werden z. B. BSA (Rinderserumalbumin) oder einfach Magermilchpulver eingesetzt. So vorbereitet kann der Western-Blot z. B. mit spezifisch gegen bestimmte Proteine gerichteten Antikörpern inkubiert werden. Die Proteine auf der Membran werden zunächst von den spezifischen Antikörpern gebunden und so durch verschiedene nachfolgende Schritte lokalisierbar (oft erst nach Einsatz eines markierten, z. B. enzymgekoppelten "sekundären" Antikörpers, der den ersten, spezifischen Antikörper erkennt). Dieses auch als Immunoblot ( vgl. Abb. ) bezeichnete Verfahren setzt voraus, daß die Proteine nach ihrer Trennung und dem Transfer auf die Membran noch ihre ursprünglichen antigenen Eigenschaften besitzen, so daß sie von den spezifischen Antikörpern erkannt werden können. Unter günstigen Umständen kann auch ein als Enzymoblot bezeichnetes Verfahren angewandt werden, bei dem die aufgetrennten und geblotteten Proteine trotz der vorausgegangenen Behandlungen so gut erhalten sind, daß Enzyme noch ihre nativen Aktivitäten besitzen. Inkubiert man dann die Membran mit für die Enzyme geeigneten Substraten, so können diese Proteine anhand der entstehenden Reaktionsprodukte, insbesondere wenn diese farbig sind, nachgewiesen werden. Eine weitere oft angewandte Methode zur Identifizierung von Proteinen nutzt deren Affinität zu bestimmten anderen Molekülen (Liganden) aus. So können radioaktiv markierte Liganden an Proteine auf der Membran binden und diese durch Autoradiographie nachweisbar machen.
Das oben beschriebene Verfahren ist auch Grundlage für die South-Western-Technik, mit deren Hilfe Protein-DNA-Interaktionen (z. B. Bindung von Transkriptionsfaktoren oder HMG-Proteinen an DNA) nachgewiesen und weiter analysiert werden können ( vgl. Abb. zur Western-Technik). Diese Technik kann als eine Synthese von Western-Technik und Southern-Technik (s. o.) angesehen werden. Bei der South-Western-Technik wird zunächst ein Western-Blot hergestellt, wobei man oft von definierten Proteingemischen, z. B. Extrakten aus Zellkernen oder Extrakten mit nachgewiesenen DNA-bindenden Aktivitäten, ausgeht. Nun wird die erhaltene Membran, nach Vorbehandlung mit geeigneten Blockierungsreagenzien, mit markierten DNA-Sonden inkubiert, die eine oder mehrere Erkennungssequenzen für DNA-bindende Proteine beinhalten. Diejenigen Proteine, welche die angebotene Sonde binden, können durch Nachweis der Markierung der Sonde (z. B. Autoradiographie bei radioaktiven Markierungen) lokalisiert und ihre relative Molekülmasse anhand von Markerproteinen bestimmt werden. Durch die Verwendung von DNA-Sonden mit unterschiedlichen Basenabfolgen können z. B. auch Affinitäten der DNA-bindenden Proteine zu bestimmten DNA-Sequenzen analysiert werden. Eine Abwandlung der South-Western-Technik wird benutzt, um Klone in einer Expressionsbibliothek zu suchen, die Proteine mit DNA-bindenden Eigenschaften produzieren (recognition site screening).
In einer der South-Western-Technik analogen Weise können mit der North-Western-Technik Protein-RNA-Interaktionen untersucht werden, indem man einen Western-Blot (mit darauf immobilisierten Proteinen) mit markierter RNA inkubiert. Hierdurch können Proteine untersucht werden, die mit RNA interagieren, wie z. B. Faktoren, die an der Translationskontrolle beteiligt sind.
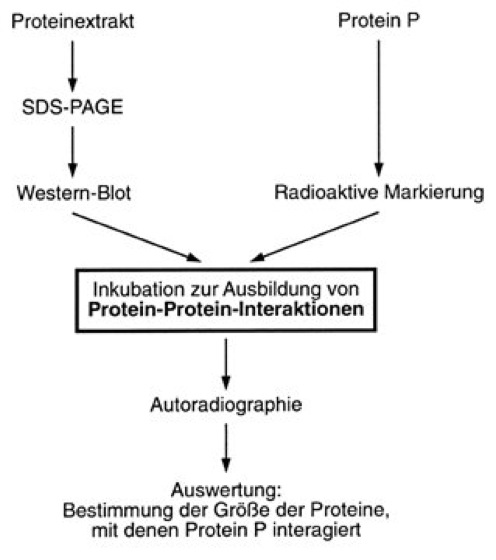
Zur Analyse von Wechselwirkungen zwischen Proteinen (Protein-Protein-Interaktionen) kann eine weitere Abwandlung der Western-Technik, die sog. Far-Western-Technik ( vgl. Abb. ), herangezogen werden. Voraussetzung für die Anwendung dieser Technik in ihrer ursprünglichen Form ist, daß spezifische Antikörper gegen das Protein, dessen Interaktionen mit anderen Proteinen untersucht werden sollen, zur Verfügung stehen. Zur Analyse der Interaktionen wird z. B. ein Proteinextrakt eines Gewebes durch SDS-Polyacrylamid-Gelelektrophorese (SDS-PAGE) aufgetrennt und wie bei der Western-Technik auf eine geeignete Trägermembran übertragen. Nach Vorbehandlungen (z. B. blocking) der Membran wird diese mit dem zu untersuchenden Protein inkubiert. Unter geeigneten Bedingungen wird dieses Protein nun an seine möglichen Interaktionspartner, die an bestimmten Stellen der Membran lokalisiert sind, binden. Die gegen das zu untersuchende Protein gerichteten Antikörper können nun, wie bei der Western-Technik, in nachfolgenden Schritten zur Lokalisierung des Proteins herangezogen werden. Neben dem reinen Nachweis einer Interaktion kann auch die Größe der interagierenden Proteine anhand von bekannten, ebenfalls aufgetrennten Markerproteinen bestimmt werden. Bisweilen kann es nötig sein, die durch die SDS-PAGE denaturierten Proteine durch bestimmte Denaturierungs- und Renaturierungstechniken wieder möglichst gut in ihre native Form zu überführen, bevor Interaktionen zwischen den Proteinen stattfinden können. Eine Vereinfachung der Far-Western-Technik kann dadurch erreicht werden, daß das Protein, dessen Interaktionen untersucht werden sollen, radioaktiv markiert wird ( vgl. Abb. ). Diese Markierung kann z. B. bei gereinigten Proteinen durch Phosphorylierung mittels spezifischer Kinasen und Einsatz von γ-32P-ATP erfolgen. Ist das zu untersuchende Protein bereits kloniert, so kann es auch durch in-vitro-Transkription und in-vitro-Translationssysteme oder unter Verwendung bakterieller Zellkulturen und von 35S-Methionin in radioaktiv markierter Form hergestellt und gereinigt werden. Wird nun dieses radioaktiv markierte Protein auf die Membran gegeben, so kann es nach erfolgter Reaktion mit seinen Interaktionspartnern ohne Einsatz von Antikörpern direkt durch Autoradiographie lokalisiert werden. Diese vereinfachte Prozedur wird zum Teil auch als Gel-overlay-Technik oder als overlay-Technik bezeichnet. Mit der Far-Western-Technik bzw. der Gel-overlay-Technik konnten z. B. Calmodulin-bindende Proteine (durch Einsatz von radioaktiv markiertem Calmodulin) oder TBP- (TATA-Box Bindeprotein) assoziierte Faktoren (sogenannte TAFs, durch Einsatz des markierten TBP; Transkriptionsfaktoren) charakterisiert werden.
Eine weitere Abwandlung der Western-Technik stellt die Shift-Western-Technik zur Analyse DNA-bindender Proteinedar. Die grundlegende Idee der blotting-Techniken, die positionsgenaue Übertragung und Immobilisierung von Makromolekülen auf eine leicht handhabbare Membran, hat auch zur Entwicklung von Techniken geführt, die nicht im eigentlichen Sinne zu den blotting-Techniken gezählt werden, da hier keine Auftrennung und zum Teil auch kein "echtes" blotten auf Membranen durchgeführt wird. Dies ist z. B. bei der slot-blot-Methode oder dot-blot-Methode der Fall, wo Protein- oder Nucleinsäuregemische direkt auf Membranen aufgetropft, dadurch immobilisiert und so auf die Anwesenheit von bestimmten Komponenten geprüft werden können. Auch der Transfer von Bakterienkolonien und Phagen-Plaques auf Membranen, der durch einfaches Auflegen der Membranen auf die Agarplatten und Abziehen der Membranen nach wenigen Minuten durchgeführt wird, gehört nicht zu den blotting-Techniken. Dieser Transfer von Bakterien (Kolonie-Lift, Kolonie-Hybridisierung) oder Phagen (Plaque-Lift, Plaque-Hybridisierung) wird z. B. bei der Suche nach bestimmten Genen oder DNA-Abschnitten in Genbibliotheken (Genbanken) durchgeführt.
E.G.
Lit.:Baldo, B.A., Tovey, E.R. (ed.): Protein Blotting. Methodology, Research and Diagnostic, Application. Basel 1989. Darling, D.C., Brickell, P.M.: Nucleinsäure-Blotting. Heidelberg, Berlin 1996. Southern, E.M.: Detection of specific sequences among DNA fragments separated by gel electrophoresis. Journal of Molecular Biology 98 (1975), 503–517.
| |
 |
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.