Lexikon der Biologie: Polymerase-Kettenreaktion

Polymerase-Kettenreaktion, polymerase chain reaction, Abk. PCR, Methode zur in-vitro-Amplifikation (Vermehrung) spezifischer DNA-Fragmente, für deren Entwicklung der amerikanische Chemiker K.B. Mullis 1993 den Nobelpreis für Chemie erhielt. Mit der überaus empfindlichen Technik läßt sich aus sehr geringen Mengen heterogener DNA (Desoxyribonucleinsäuren) innerhalb weniger Stunden ein bestimmtes doppelsträngiges DNA-Fragment (Doppelstrang) millionenfach amplifizieren (anreichern).
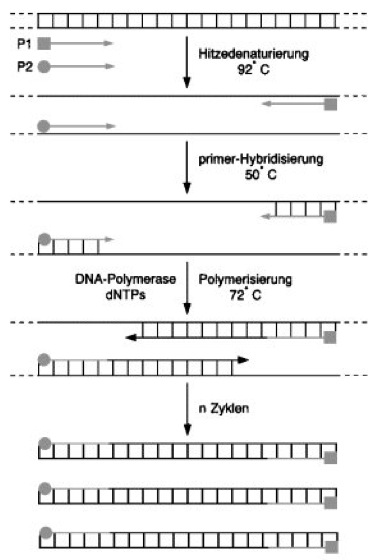
Der Reaktion liegt folgendes Prinzip zugrunde (vgl. Abb. 1): DNA, welche die zu vermehrende Sequenz (Nucleotidsequenz) enthält, wird durch Hitze (ca. 92 °C) in ihre Einzelstränge zerlegt. Durch Temperaturerniedrigung auf ca. 50 °C werden 2 chemisch synthetisierte Oligodesoxynucleotide (primer) an die denaturierte DNA (Denaturierung) anhybridisiert (Hybridisierung). Die Sequenz dieser primer ist so gewählt, daß sie komplementär (Komplementarität) zu jeweils einem der Bereiche ist, die die zu vermehrende DNA flankieren. Die so entstehenden kurzen doppelsträngigen DNA-Regionen mit den in Richtung der zu amplifizierenden DNA weisenden 3'OH-Enden der primer sind Substrat für DNA-Polymerase. Unter für das Enzym geeigneten Temperaturbedingungen werden die primer mit dNTPs (2'-Desoxyribonucleosid-5'-triphosphate) verlängert, so daß die entstehenden DNA-Stränge nun als Matrize für den jeweils anderen primer dienen können. Wiederholte Hitzedenaturierung der DNA-Doppelstränge, Anhybridisieren der primer und Verlängerung durch DNA-Polymerase (als ein Temperaturzyklus bezeichnet) führt zur enormen Anreicherung eines doppelsträngigen (ds-) DNA-Fragments, das von den 5'-Enden der primer begrenzt wird.
Nach 20 Zyklen erhält man von einem DNA-Molekül ausgehend infolge einer exponentiellen Kettenreaktion ca. 1 Million (220) Kopien. Genetisches Material, welches nur in Spuren vorhanden ist (s.u.), kann mit Hilfe der PCR einer molekularbiologischen Analyse zugänglich gemacht werden. Eine breite Anwendung dieser Methode wurde 1988 durch Einführung der Taq-DNA-Polymerase, eines thermostabilen Enzyms, eingeleitet. Inzwischen sind weitere hitzeresistente DNA-Polymerasen isoliert worden. Da diese Enzyme im Gegensatz zur bis dahin verwendeten Klenow-Polymerase (Klenow-Fragment) durch den Denaturierungsschritt nicht inaktiviert werden und damit eine wiederholte Zugabe in den Reaktionsansatz entfällt, wird eine Automatisierung des Amplifikationsverfahrens ermöglicht. Mit Hilfe von Heizelementen, die imstande sind, in definierten Zeitabständen die Temperatur zu wechseln, benötigt man für eine Abfolge von 20 Temperaturzyklen ca. 2 h.
Das einfache Konzept der PCR hat seit ihrer Einführung Mitte der 1980er Jahre eine Vielzahl methodischer Varianten erfahren. Mit primern, die an ihrem 5'-terminalen Ende eine zur Zielsequenz nicht-komplementäre Erweiterung aufweisen, lassen sich endständig Fremdsequenzen mitamplifizieren. Diese können zum einen Erkennungssequenzen für Restriktionsendonucleasen (Restriktionsenzyme) sein, welche eine anschließende Klonierung des Amplifikationsprodukts in einen Vektor erleichtern, zum anderen aber auch Promotor-Erkennungssequenzen, die eine Transkription des Amplifikats ermöglichen. Einzelne Abweichungen innerhalb der primer-Sequenzen zur Ziel-DNA bieten die Möglichkeit einer in-vitro-Mutagenese (vgl. Abb. 2) der Amplifikate. Diese Abweichungen können sowohl nicht-komplementäre Basen als auch Insertionen bzw. Deletionen sein.
Bei der sog. error prone PCR wird hingegen die natürliche Fehleinbaurate der zur Amplifikation verwendeten DNA-Polymerase durch bestimmte Reaktionsbedingungen erhöht, wodurch die entstehenden Amplifikationsprodukte an zufälligen Positionen Sequenzabweichungen zur Ausgangs-DNA aufweisen. Diese Methode wird u.a. bei in-vitro-Evolutions-Experimenten (in-vitro-Evolution) eingesetzt, bei denen durch gerichtete Selektion, Amplifikation und Mutation im Reagenzgefäß Nucleinsäuren mit bestimmten Eigenschaften erzeugt werden können.
Bei der asymmetrischen PCR ( vgl. Abb. 3 ) ist einer der Amplifikations-primer limitierend im Reaktionsansatz vorhanden. Ist dieser aufgebraucht, werden nur noch Amplifikationsprodukte mit Hilfe des überschüssigen primers synthetisiert. Auf diese Weise läßt sich einzelsträngige DNA, wie sie u.a. zur Sequenzierung oder auch als Hybridisierungssonde benötigt wird, anreichern. Eine Markierung von DNA-Fragmenten kann durch Einsatz markierter primer bzw. dNTPs erreicht werden.
Die als inverse PCR bezeichnete Methode ( vgl. Abb. 4 ) ermöglicht die Amplifikation zweier unbekannter DNA-Abschnitte, die durch eine bekannte DNA-Sequenz getrennt sind. Mittels RT-PCR (Abk. für reverse transcription-PCR) können auf indirektem Weg auch RNA-Moleküle (Ribonucleinsäuren) „amplifiziert“ werden, wodurch man mit geringem Zeitaufwand zu Aussagen über den Expressionszustand einzelner Gene (Genexpression) gelangen kann. In einem ersten Schritt findet dabei ein Umschreiben der RNA in cDNA durch das Enzym reverse Transkriptase statt. Dies kann entweder mit einem Transkript-spezifischen primer durchgeführt werden, oder aber man benutzt, um einen Pool sämtlicher cDNAs anzulegen, in ihrer Sequenz zufällige Hexanucleotid-primer (random-primer, s.u.). Die entstehende cDNA, die im Gegensatz zu RNA von thermostabilen DNA-Polymerasen als natürliches Substrat akzeptiert wird, kann anschließend in einem zweiten Schritt in einer PCR eingesetzt werden. Inzwischen sind jedoch auch thermostabile DNA-Polymerasen isoliert worden, welche unter bestimmten Reaktionsbedingungen Reverse-Transkriptase-Aktivität aufweisen. Unter Einsatz dieser Enzyme in RT-PCRs sind nach erfolgter reverser Transkription lediglich eine Änderung in die für die DNA-Synthese optimalen Reaktionsbedingungen sowie die Zugabe von Amplifikations-primern notwendig. Ein weiterer Vorteil dieser Enzyme besteht darin, daß die reverse Transkription bei hohen Temperaturen durchgeführt werden kann, wodurch die Ausbildung von Sekundärstrukturen der RNA, die unter Umständen zu einem Abbruch der cDNA-Synthese führen würden, vermindert wird.
Unter kompetitiver PCR versteht man eine Variante der RT-PCR, mit der quantitativ die Menge eines bestimmten Transkripts bestimmt werden kann. Nach reverser Transkription wird eine bekannte Konzentration eines DNA-Moleküls hinzugefügt, welches die gleiche primer-Bindesequenz wie die zu analysierende cDNA besitzt, sich jedoch in der Länge vom erwarteten cDNA-Amplifikationsprodukt unterscheidet. Bei der anschließenden PCR konkurrieren die Proben-cDNA und der DNA-Standard bezüglich der Bindewahrscheinlichkeit um die Amplifikations-primer (Kompetition). Die Produktmenge des DNA-Standards im Vergleich zur Produktmenge der cDNA-Probe kann als Maß für die Menge analysierter RNA verwendet werden (quantitative PCR, s.u.).
Die sog. anchored PCR kann zur Amplifikation von unbekannten Regionen, die 5'- bzw. 3'-terminal von bereits bekannten Sequenzen liegen, angewandt werden. Das Prinzip dieser Variante der PCR beruht darauf, bekannte Sequenzen (sog. Anker, anchor) flankierend zu noch unbekannten Sequenzbereichen zu erzeugen. Praktisch geschieht dies z.B. durch Anhängen eines Oligonucleotids an die zu analysierende Nucleinsäure. Eine zu Beginn der Arbeit bekannte Sequenz sowie die angehängte Oligonucleotidsequenz werden zur Auswahl von primern benutzt, mit denen man in einer PCR den unbekannten Zwischenbereich amplifizieren kann.
Die multiplex-PCR, bei der, im Gegensatz zu einer üblichen PCR, mehrere primer-Paare in einem Reaktionsansatz eingesetzt werden, wird vor allem in der genetischen Diagnostik verwendet (s.u.). Unter Anwendung der multiplex-PCR lassen sich mehrere PCRs simultan mit einer zu untersuchenden Probe durchführen. Die primer-extension-preamplification dient dazu, nur in geringen Mengen vorhandenes genetisches Material vor der eigentlichen PCR mit spezifischen primern unspezifisch zu vervielfältigen, um mehrere PCR-Analysen möglich zu machen. Die zu analysierende DNA wird dabei in einer ersten Amplifikationsrunde mit einem Gemisch sehr vieler verschiedener, kurzer, in ihrer Sequenz zufälliger primer (random-primer; random priming, random-PCR) versetzt, wodurch das Erbmaterial millionenfach vervielfältigt wird. Ein Bruchteil dieser Amplifikationsprodukte kann dann für eine zweite PCR mit spezifischen primern verwendet werden.
Eine Kombination aus in-situ-Hybridisierung und PCR stellt die in-situ-PCR dar. Bei dieser Methode werden in Paraffin eingebettete Gewebeschnitte auf mit Bindesilan vorbehandelten Objektträgern fixiert und anschließend einer Proteasebehandlung unterzogen. Der Objektträger mit freigelegten Nucleinsäuren wird dann mit Amplifikationslösung überschichtet und auf dem Thermoblock eines PCR-Geräts befestigt. Bei 60 °C (hot start) wird thermostabile DNA-abhängige DNA-Polymerase zugegeben, der Gewebeschnitt mit einem Deckglas bedeckt und mit Paraffinöl überschichtet. Anschließend wird die PCR durchgeführt. Die Sichtbarmachung der gewünschten Nucleinsäure kann nach Beendigung der Amplifikationsreaktion entweder indirekt über eine nachfolgende normale in-situ-Hybridisierung oder, falls Digoxigenin-dUTP (Digoxigenin) in der Amplifikationsreaktion verwendet wurde, direkt in einem Immunassay erfolgen. Die in-situ-PCR bietet aufgrund höherer Nachweisempfindlichkeit gegenüber der in-situ-Hybridisierung die Möglichkeit, die Verteilung bestimmter Nucleinsäuren, z.B. den Werdegang viraler DNA bzw. RNA in einer Zelle, zu beobachten.
Eine Variante der PCR mit erhöhter Sensitivität ist die sog. nested PCR („ineinandergeschachtelte PCR“). Eine hochempfindliche Methode zum Nachweis von Antigenmolekülen stellt die Immuno-PCR dar. Diese Methode kombiniert die auf der Antikörperbindung basierende spezifische Epitop-Erkennung mit der enormen Amplifikationskapazität der PCR. Eine weitere Variante der PCR ist die sog. Blasen-Linker-PCR. Durch die sog. quantitative PCR läßt sich die die Ausgangsmenge zur PCR verwendeter DNA- bzw. cDNA-Moleküle berechnen.
Außerhalb der Grundlagenforschung findet die PCR vor allem in der medizinischen Diagnostik (Biomedizin, biomedizinische Technik, Medizin, molekulare Medizin) praktische Anwendung. Sowohl bei der pränatalen genetischen Diagnose (pränatale Diagnostik), bei der in der Regel nur wenig Zellmaterial zur Verfügung steht, als auch bei der Virusdiagnostik (Viruskrankheiten), wo sich mit Hilfe der PCR schon sehr früh, noch bevor Antikörper gegen das Virus gebildet werden, eine Infektion (Virusinfektion) nachweisen läßt, hat sich die PCR inzwischen als äußerst hilfreich erwiesen. Der Evolutionsbiologie ist durch die PCR eine genetische Analyse paläontologischer Funde (Fossilien, Koprolithen, Paläontologie) ermöglicht worden (molekulare Archäologie, molekulare Paläontologie). So konnten z.B. DNA-Fragmente aus den Überresten einer 7000 Jahre alten indianischen Leiche amplifiziert, sequenziert und einer Homologisierung mit DNA (Sequenzhomologie) heute lebender Bevölkerungsgruppen unterzogen werden. Auch in der Kriminalistik findet die PCR Anwendung. Geringste Mengen an Blut oder Sperma können, nach Amplifikation und Charakterisierung der darin enthaltenen Nucleinsäure, zur Identifizierung eines Straftäters beitragen.
Amplikon, Amplimere, ASO, binding site selection assay, Biochemie (Geschichte der), CASTing, Chorionzottenbiopsie, chromosome crawling, Competitor-Nucleinsäuren, Differential Display, DNA-fingerprinting (Abb.), DNA-Synthesizer, forensische Biochemie, forensische Gentechnik (Abb.), Genamplifikation, Gendatei, genetischer Fingerabdruck, Genomanalyse, genome walking, Genomik, Gentechnologie, HIV-Test, Immun-Krebstherapie, kurze Matrize, lange Matrize, Mikroreaktoren, molekulare Ökologie, Novel Food, Oligonucleotidsynthese, Phagen-Display-Bibliothek, Taxonomie, Zahnpulpa-DNA.
R.M.
Lit.:Müller, H.-J.: Polymerase-Kettenreaktion (PCR) – Das Methodenbuch. Heidelberg 2001. Mullis, K.B., Ferré, F., Gibbs, R.A. (eds.): The polymerase chain reaction. Basel 1994. Newton, C.R., Graham, A.: PCR. Heidelberg 21997. Rabinow, P.: Making PCR: A Story of Biotechnology. Chicago IL 1996.
Polymerase-Kettenreaktion
Abb. 1:Prinzip der PCR: DNA, die das zu amplifizierende Fragment enthält, wird durch Hitze (92 °C) denaturiert. Zwei primer P1 und P2, die eine komplementäre Sequenz zu den Bereichen aufweisen, welche die Ziel-DNA flankieren, werden durch Abkühlen des Reaktionsansatzes auf ca. 50 °C an diese anhybridisiert. Durch Temperaturerhöhung auf 72 °C wird die Verlängerung der primer mit dNTPs durch eine thermostabile DNA-Polymerase (z.B. Taq-DNA-Polymerase) eingeleitet. Mehrfache Wiederholung dieses Temperaturzyklus führt zur Anreicherung des durch die primer P1 und P2 eingegrenzten DNA-Fragments.

Polymerase-Kettenreaktion
Abb. 2:In-vitro-Mutagenese bzw. Ankoppeln einer Fremdsequenz mittels PCR:
Durch zur Zielsequenz nicht komplementäre Basen innerhalb eines primers (P1) können durch die PCR DNA-Fragmente erzeugt werden, welche im Randbereich eine zur Originalsequenz abweichende Base enthalten. Des weiteren können durch eine 5'-terminale Erweiterung eines primers (P2) zur Zielsequenz nichtkomplementäre Fremdsequenzen (Restriktionsschnittstellen, Promotor-Erkennungssequenzen) gezielt an DNA-Fragmente angekoppelt werden.
Polymerase-Kettenreaktion
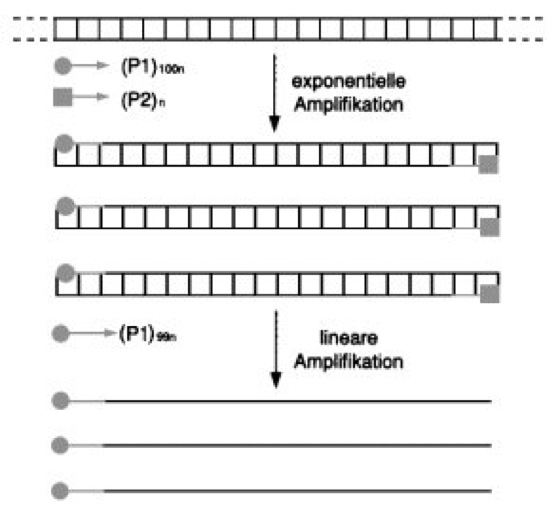
Abb. 3:Asymmetrische PCR: Zur Erzeugung definierter einzelsträngiger DNA-Fragmente wird dem Reaktionsansatz ein primer (P1) gegenüber dem zweiten primer (P2) im ca. 100molaren Überschuß zugegeben. Ist P2 nach exponentieller Amplifikation des durch die primer P1 und P2 eingegrenzten DNA-Bereichs aufgebraucht, werden mit dem noch vorhandenen primer P1 linear DNA-Einzelstränge angereichert.

Polymerase-Kettenreaktion
Abb. 4:Inverse PCR: Durch Restriktionsschnitt mit Enzym R1 wird ein definiertes DNA-Fragment erzeugt, welches den in seiner Sequenz bekannten Anteil X, flankiert von den beiden unbekannten Sequenzabschnitten Y und Z, enthält. Nach Zirkularisierung dieses Fragments mit DNA-Ligase und einem weiteren Restriktionsschnitt mit R2 kann mit den primern P1 und P2 ein DNA-Fragment amplifiziert werden, das die beiden unbekannten Sequenzabschnitte Y und Z enthält.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.