Lexikon der Biochemie: Oligonucleotidsynthese
Oligonucleotidsynthese, die chemische Synthese von Oligonucleotiden durch Knüpfung einer Phosphodiesterbindung zwischen der 5'-Hydroxy-Gruppe des einen Nucleotids und der 3'-Phosphat-Gruppe des anderen Nucleotids oder umgekehrt zwischen einer 3'-Hydroxygruppe und einem 5'-Phosphat zweier Nucleotide. Für präparative O. wird wegen der höheren chemischen Reaktivität der primären 5'-Hydroxygruppe fast ausschließlich die zuerst genannte Reaktion verwendet. Alle funktionellen Gruppen, die während der Knüpfung der Internucleotidbindung unerwünschte Nebenreaktionen eingehen können, müssen durch Einführung selektiv abspaltbarer Schutzgruppen reversibel blockiert werden. Bei der Synthese von Oligoribonucleotiden muss auch die zusätzliche 2'-Hydroxygruppe geschützt werden.
Prinzipiell unterscheidet man bei der O. zwischen der Phosphodiester-Methode (Abb. 1a) und der Phosphotriester-Methode (Abb. 1b), die sich vor allem darin unterscheiden, dass Triester wegen der fehlenden Ladung nicht mehr polar sind, sich gut in unpolaren Solvenzien lösen und auch keine unerwünschten Reaktionen mit bestimmten Aktivierungsmitteln eingehen. Obgleich mit der Diestermethode Oligonucleotide der Kettenlänge 10-20 synthetisiert werden konnten, ist dieses Verfahren für den Aufbau von Gensegmenten weniger gut geeignet (u.a. niedrige Ausbeuten; lange Reaktionszeiten; ungeschützte Phosphatgruppe in der zwischen zwei Nucleosiden gebildeten Phosphodiesterbindung). Die effektivsten Syntheseverfahren basieren auf der Triestermethode bzw. auf abgeleiteten Varianten unter Einbeziehung der Merrifield-analogen Polymersynthese (Festphasen-Peptidsynthese) am festen Träger.
Die reaktiven Gruppen der Mononucleoside werden durch Schutzgruppen reversibel blockiert, um unerwünschte Nebenreaktionen weitgehend zu vermeiden und die Synthese in die gewünschte Richtung zu drängen. Als Schutzgruppe (R1) für die primäre Hydroxyfunktion fungiert die mit aromatischen Sulfonsäuren, Trifluoressigsäure oder ZnBr2 abspaltbare Dimethoxytrityl- (DMTr-) Gruppe, während für die sekundäre 3'-Hydroxyfunktion vorrangig basenlabile Acetyl- bzw. Benzoylreste (R2) eingesetzt werden. Als N-Schutzgruppen der Nucleobasen (B) werden der Anisoylrest für Cytosin, der Benzoylrest für Adenosin und der Isobutyrylrest für Guanosin bevorzugt, die gewöhnlich durch Ammonolyse entfernt werden. Wichtige Phosphatschutzgruppen (R3) sind die durch β-Eliminierung abspaltbare β-Cyanoethylgruppe sowie insbesondere chlorsubstituierte Phenylester mit verschiedenen Deblockierungsmöglichkeiten.
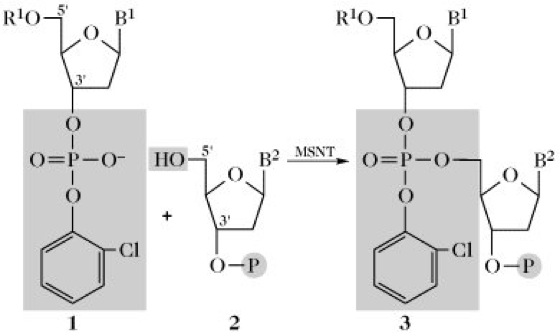
Kondensationsmittel (Aktivierungsmittel) für die Knüpfung der Internucleotidbindung sind abhängig von der gewählten Synthesestrategie. Während sich Dicyclohexylcarbodiimid (DCC), Mesitylensulfonylchlorid (MS) und Triisopropylbenzolsulfonylchlorid (TPS) als effektiv bei der Diestermethode erwiesen haben, wird für die Triestermethode bevorzugt 1-(Mesitylen-sulfonyl)-3-nitrotriazol (MSNT) verwendet.
Trotz der Vielzahl bekannter Varianten der O. werden in der Praxis vorrangig die beiden nachfolgenden Verfahren benutzt:
1) Phosphotriester-Polymersynthese (Abb. 2). Die Phosphatkomponente (1) wird als ein in Pyridin lösliches Triethylammoniumsalz des Phosphodiesters eingesetzt. Sie reagiert mit der trägergebundenen Nucleosidkomponente in Gegenwart des Kupplungsreagens MSNT zum polymergebundenen, geschützten Dinucleotid (3). Bei nicht vollständiger Umsetzung der Hydroxykomponente (2) wird die 5'-Hydroxyfunktion acetyliert (capping-Schritt), um eine Bildung von Fehlsequenzen zu verhindern. Danach wird die Schutzgruppe R1 von der Verbindung 3 abgespalten, und es beginnt die Anknüpfung des nächsten partiell geschützten Nucleotids. Mit MSNT als Aktivierungsmittel betragen die Kupplungszeiten je Kupplungsschritt 45min bei 45°C. Ein Zyklus erfordert etwa 60 min. Für den Aufbau längerer Oligonucleotide setzt man zweckmäßigerweise entsprechend geschützte Dimere oder Trimere, die nach der Triestermethode in Lösung hergestellt werden, als Phosphatkomponenten ein, wodurch die Synthesedauer drastisch verkürzt wird. Als polymere Träger werden Copolymerisate aus Polystyrol und 1% Divinylbenzol verwendet, aber auch Polyamide, Kieselgel, Glasperlen oder Cellulosepapier. Analog zur Peptidsynthese am polymeren Träger stehen verschiedene kommerzielle, halbautomatische wie auch programmgesteuerte DNA-Synthesizer zur Verfügung. Mit den vollautomatisch arbeitenden Syntheseautomaten dauert die Verknüpfung zweier Nucleotide einschließlich aller Nebenreaktionen weniger als 15min. Die Reaktionsfolge Kupplung und selektive Abspaltung der 5'-Hydroxyschutzgruppe wird so lange wiederholt, bis die gewünschte Sequenz am Träger synthetisiert ist. Danach werden alle Schutzgruppen sowie der polymere Träger abgespalten, und das Endprodukt wird durch HPLC oder Polyacrylamid-Gelelektrophorese gereinigt. Die Bestätigung der Identität des Syntheseprodukts erfolgt durch Sequenzanalyse oder durch einen zweidimensionalen Fingerprint, d.h. zweidimensionale Trennung der Hydrolyseprodukte nach Inkubation mit Schlangengift-Diesterase.
Eine Alternative zu den relativ teuren Syntheseautomaten ist die sog. Filterblättchen-Methode, bei der die Startnucleotide an geeignete Papierfilter gebunden werden. Wenn man die Kondensationsreaktionen mit jedem einzelnen Nucleotid in getrennten Reaktionsgefäßen durchführt und die Filter nach jedem Syntheseschritt entsprechend des vorgegebenen Syntheseplans in die entsprechenden Reaktionsgefäße umsortiert (da sie wie Teebeutel eingehängt werden, spricht man im Laborjargon auch von der tea-bag-Methode), kann man gleichzeitig viele Oligonucleotide synthetisieren.
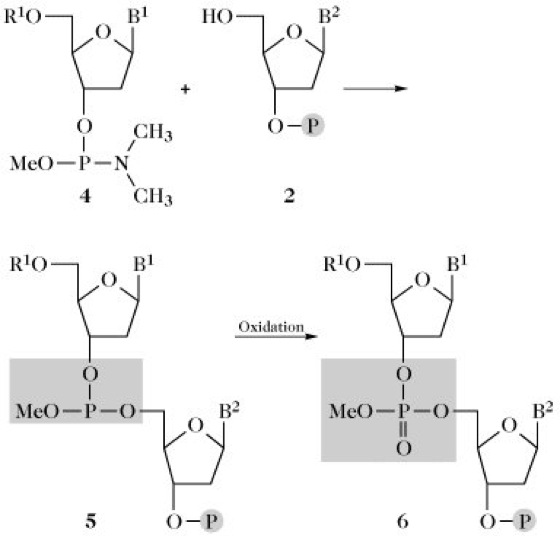
2) Phosphit-Triester-Polymersynthese (Abb. 3). Grundlage dieses Verfahrens ist die Phosphitmethode, bei der zunächst ein Nucleotidphosphitester (5) gebildet wird, der anschließend mit Iod zum Phosphotriester oxidiert wird. Die partiell geschützten Desoxynucleosid-Phosphoramidite (4) (Phosphoramidit-Methode), dargestellt aus dem entsprechenden Nucleosid und Chloro(N,N-dimethylamino)-methoxyphosphan, CH3O-P(Cl)-N(CH3)2, sind äußerst reaktiv und ermöglichen nahezu quantitative Umsätze bei Kupplungszeiten von nur 5min. Diese hochreaktiven Reagenzien (Reaktionsbomben) stellen in Verbindung mit Injektionsspritze und Serumkappe das Handwerkszeug des modernen Nucleinsäuresynthetikers dar. Die Dauer eines Reaktionszyklus wird mit etwa 30 min angegeben. Als polymerer Träger fand unter anderem Kieselgel Verwendung. Nach der Synthese werden die Schutzgruppen abgespalten und das Oligonucleotid vom Träger abgelöst. Die Reinigung erfolgt wie unter 1) beschrieben, wofür zusätzlich Reversed-Phase-Chromatographie und Gelchromatographie wichtig sind. Unter den besten Bedingungen kann man heute Oligonucleotide bis zu einer Länge von etwa 200 Basen in einem Stück synthetisieren.
Die beiden Synthesevarianten am polymeren Träger erlauben einen schnellen Aufbau von Oligonucleotiden, die Ausgangsprodukte für die Gensynthese darstellen, aber auch für andere molekularbiologische Aufgabenstellungen Verwendung finden, z.B. als Sonden für die Isolierung von mRNA, als Primer für die Sequenzierung von DNA (oder mRNA) und für die Polymerase-Kettenreaktion, als Hybridisierungsproben und Linkersequenzen für gentechnische Operationen.
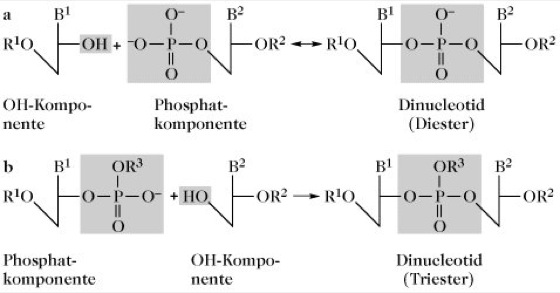
Oligonucleotidsynthese. Abb. 1. a) Phosphodiester-Methode, b) Phosphotriester-Methode.
Schreiben Sie uns!