Lexikon der Biochemie: Ribosomen
Ribosomen, Monosomen, zelluläre Orte der Proteinbiosynthese mit Multienzymcharakter. Es sind rundliche bis ellipsoide, stark hydratisierte Zellorganellen von 15-30 nm Durchmesser, die normalerweise im Cytoplasma als Polyribosomen vorliegen. R. wurden erstmals 1953 von Palade (Nobelpreis 1974) beschrieben.
Der Ribosomengehalt einer Zelle ist direkt mit deren Proteinsyntheseaktivität korreliert. Nach Vorkommen und Größe kann man zwei Hauptklassen von R. unterscheiden (Tab.): 80S-R. im Cytoplasma aller eukaryontischen Zellen, 70S-R. in prokaryontischen Zellen, Plastiden und in einem Teil der Mitochondrien; die Mitochondrien der Wirbeltiere enthalten 55S-R (Farbtafel).
Die Untereinheitenzusammensetzung der R. wird wesentlich von der Ionenkonzentration des Suspensionsmediums bestimmt, insbesondere durch den Mg2+-Gehalt. Wird dieser auf <0,001M erniedrigt, dissoziieren die R. in zwei morphologisch wie funktionell ungleiche Untereinheiten (Abb. 1). Die 70S-R. bestehen dann aus einer großen 50S- und einer kleinen 30S-Untereinheit, während sich die 80S-R. aus 60S- und 40S-Untereinheiten zusammensetzen. Durch völligen Mg2+-Entzug oder durch Zusatz von etwa 1M Lösungen monovalenter Kationen, wie Li+, Cs+, K+, zerfallen die Untereinheiten in noch kleinere diskrete Partikel, die Ribonucleoproteinkerne oder core-Partikel (engl. core Kern). Außerdem werden bestimmte split-Proteine abgelöst (engl. split, Abspaltung). Die core-Partikel und split-Proteine weisen keine Translationsaktivität mehr auf, können sie jedoch durch Reassoziation der Komponenten wieder zurückgewinnen.
Zusammensetzung und Feinstruktur der R. (Farbtafel). Beide Untereinheiten des E.-coli-R. sind asymmetrisch. Die große (50S-) Untereinheit besitzt einen zentralen Höcker (Kopf) und zwei seitliche Arme (den gefurchten und den L7/L12-Stiel), die auf beiden Seiten des Kopfs in einem Winkel von 50° geneigt sind. Die kleine (30S-) Untereinheit enthält eine Spalte bzw. Einkerbung, die die Struktur in zwei ungleiche Teile teilt. Dadurch entstehen folgende Bereiche: die Plattform, der Kopf und der Sockel. Im intakten R. stehen die Plattform und ein großer Teil des Sockels der 30S-Untereinheit mit der 50S-Untereinheit in Kontakt.
Im Verlauf der Evolution blieb die Ribosomenstruktur hoch konserviert und ist auch für die meisten entfernt verwandten Organismen ähnlich. Die Elektronenmikroskopie offenbart allerdings geringe, jedoch deutliche Unterschiede in den Ribosomenformen von Eubakterien, Archaebakterien, Eocyten und Eukaryonten (cytoplasmatische R.), die zur Interpretation phylogenetischer Verwandtschaftsbeziehungen zwischen diesen Gruppen herangezogen wurden. [J.A. Lake Ann. Rev. Biochem. 54 (1985) 507-530]
Die R. bestehen lediglich aus RNA (ribosomale RNA) und Proteinen (Ribosomenproteine). 70S-R. enthalten 65% RNA und 35% Protein, 80S-R. je 50 % RNA und Protein. Die R.-RNA (rRNA) weist etwa 70 % helicale Abschnitte auf, die mit nichthelicalen Bereichen abwechseln. Letztere sind durch Ionen- und Wasserstoffbrückenbindungen mit den Ribosomenproteinen in Form spezifischer Nucleotid-Aminosäure-Wechselwirkungen miteinander verknüpft (Abb. 2).
Die Totalrekonstitution der Ribosomenuntereinheiten aus isolierten und aufgetrennten RNA- und Proteinarten ist Nomura und Mitarbeitern zwischen 1969 und 1972 gelungen. Die Reassoziation verläuft spontan und kooperativ durch Selbstorganisation, d.h., alle Informationen, die für eine korrekte Assoziation eines R. benötigt werden, sind in der Struktur seiner Komponenten enthalten. Die Rekonstitution der 30S-Untereinheit vollzieht sich bei 40 °C bei hohen KCl-Konzentrationen und dauert ungefähr 10 Min. Die Rekonstitution der 50S-Untereinheit verläuft langsamer und erfordert höhere Temperaturen. Über die Rekonstitution eukaryontischer Ribosomen liegen keine Veröffentlichungen vor. In der Zelle werden die Ribosomenuntereinheiten zuerst im Nucleolus aus rRNA und Proteinen zusammengesetzt und dann in das Cytoplasma exportiert.
Ribosomen. Tab. Vergleich einiger Eigenschaften von 70S- und 80S-Ribosomen.
| |||
| Mr (× 106Da) | 2,7 | 4,0 | |
| S-Werte der Unterein-heiten | 50 + 30 | 60 + 40 | |
| %RNA | 65 | 50 | |
| S-Werte der hochmole-kularen rRNA | 23 + 16 | 28 + 18 | |
| Mr der hochmolekularen rRNA (× 106Da) | 1,1 + 0,56 | 1,7 + 0,7 | |
| GC-Gehalt der hoch-molekularen rRNA (%) | 54 + 54 | 67 + 59 | |
| Anzahl der Ribosomen-proteine | 34 + 21 | ungefähr 70 | |
| Initiation der Proteinbio-synthese durch | Formyl-Met-tRNAF | Met-tRNAMet | |
| Inhibierung der Protein-biosynthese: | |||
| durch Chloramphenicol, durch Cycloheximid | + – | - + |

Ribosomen. Abb. 1. Schematische Darstellung eines Ribosoms. AS = Aminosäure.
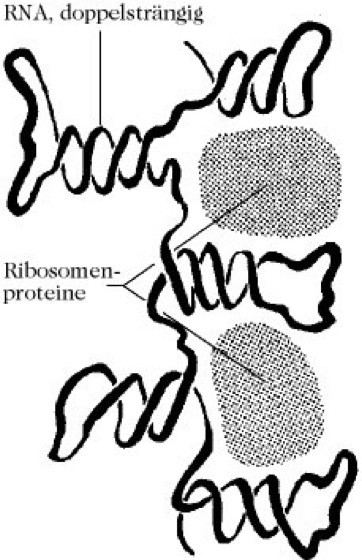
Ribosomen. Abb. 2. Schematische Darstellung der Anordnung von ribosomaler RNA und Ribosomenproteinen im Ribosom (Farbtafel).
Schreiben Sie uns!