News: Dem Fehler auf der Spur
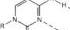
Daher ersann Richard Hamming von den Bell Telephone Laboratories bereits im Jahr 1950 eine elegante Methode, um Fehler bei der Übertragung digitaler Daten sofort zu erkennen. Dabei wird am Ende jeder Nachricht, die ja lediglich aus Nullen und Einsen besteht, das so genannte Paritätsbit angehängt, sodass die Quersumme immer eine gerade Zahl ergibt: Die Botschaft 100110 erhält das Extrabit 1, sodass sie sich in 100110,1 verwandelt, die Nachricht 100001 wird dagegen um eine zusätzliche 0 als Prüfbit bereichert (100001,0). Verändert ein Übertragungsfehler eine einzige Ziffer, dann wird die Quersumme ungerade – die Botschaft kann ohne aufwändige Entschlüsselung gleich als falsch verworfen werfen.
Was hat das mit dem Erbmolekül, der DNA, zu tun? Sehr viel, sagt Dónall Mac Dónaill vom Trinity College Dublin. Der Chemiker fragte sich, warum das Alphabet des Lebens lediglich aus Nucleotiden mit den vier Basen Adenin (A), Thymin (T), Guanin (G) und Cytosin (C) besteht. Schließlich gibt es noch weitere insgesamt 16 Basen, die teilweise in RNA-Molekülen auftauchen und hier ebenfalls ihre Funktion erfüllen. Bisher gingen Molekularbiologen davon aus, dass lediglich der Zufall die vier Nucleotide aus der Ursuppe zu Beginn des Lebens fischte und damit die DNA aufbaute.
Doch Mac Dónaill sieht hier ebenfalls ein Paritätsbit am Werk, das Fehler erkennen soll. Er interpretierte die Basenpaarung der DNA als drei-Ziffern-Code: So ist Guanin mit Cytosin über drei Wasserstoffbrücken verknüpft, wobei die Base Guanin zwei Protonendonatoren und einen Protonenakzeptor, die Base Cytosin dagegen zwei Protonenakzeptoren und einen Protonendonator beisteuert. Umgesetzt in Binärcode lautet der Schlüssel für das Nucleotid Guanin 011, für Cytosin dagegen 100. Erweitert um ein Prüfbit, verwandelt sich Guanin in 011,0 und Cytosin in 100,1 – die gesamte Quersumme ist jeweils gerade. Auch die Basen Adenin und Thymin, bei denen ebenfalls jeweils drei Protonendonatoren beziehungsweise -akzeptoren vorliegen, die allerdings nur zwei Wasserstoffbrücken bilden, können mit solchen Paritätsbits ergänzt werden.
Die Zelle müsste nach der Vorstellung von Mac Dónaill jetzt nur noch prüfen, ob die Quersumme gerade oder ungerade ist. Paart sich beispielsweise Guanin (011,0) mit einem Nucleotid, dessen Ziffernfolge 010,0 lautet, dann wäre die Quersumme ungerade – die Paarung könnte als falsch erkannt werden.
"Das ist eine neue Idee, die andere dazu anregen sollte, weitere Aspekte aus der Informatik im genetischen Code aufzuspüren", meint der Computerspezialist und Chemiker Graham Richards von der Oxford University. "Instinktiv würde man annehmen, dass der DNA-Code ein System entwickelt hat, das Fehler minimiert. Mac Dónaills Arbeit zeigt, wie das erreicht worden sein könnte."

Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.