Lexikon der Chemie: Proteine
Proteine, Eiweißstoffe, ausschließlich oder überwiegend aus Aminosäuren aufgebaute makromolekulare Verbindungen, die als Biopolymere entscheidender Bestandteil der lebenden Materie sind. In einer Escherichia-coli-Zelle sind 3000 verschiedene P. enthalten, mehr als 100000 unterschiedliche P. finden sich im menschlichen Organismus. P. bestimmen Struktur und Funktion jeder Zelle. Als Enzyme und Peptidhormone sind sie für den geregelten Ablauf der chem. Reaktionen des Stoffwechsels verantwortlich. Als Strukturproteine (Gerüstproteine, Skleroproteine), z. B. Kollagene, Elastine, Keratine, sind sie wesentlicher Bestandteil von Stützgewebe, Bindegewebe und Biomembranen. Als kontraktile P., z. B. Actin und Myosin, ermöglichen sie den Kontraktionsprozeß der Muskelfaser. Als Immunglobuline oder Interferone bilden sie spezifische körpereigene Abwehrproteine. Als Carrierproteine, z. B. Hämoglobine, Cytochrome, Transferrin, Coeruplasmin, sind sie an Elektronenübertragungsprozessen der Photosynthese und Atmung beteiligt oder transportieren Stoffwechselprodukte und Metall-Ionen. Als Speicherproteine, z. B. Eialbumine, Milchcasein, sichern sie die Aminosäurereserve des Organismus, als Rezeptorproteine vermitteln sie die spezifische Bindung von Wirkstoffmolekülen am Wirkort. Darüber hinaus sind P. beim Blutgerinnungsprozeß, bei der Blutgruppenspezifizierung, bei der Steuerung der Genaktivitäten und bei der Regulation vieler anderer biochem. Prozesse von entscheidender Bedeutung.
Einteilung. Nach dem Vorkommen in Organismen unterscheidet man pflanzliche und tierische P., Virusproteine, Bakterienproteine, nach dem Vorkommen in Organen und Zellorganellen z. B. Plasma-, Muskel-, Milch-, Eiproteine sowie Ribosomen-, Zellkern-, Mikrosomen- und Membranproteine. Nach der allgemeinen biologischen Funktion lassen sich die P. in Enzym-, Struktur-, Transport-, Speicher- und Rezeptorproteine unterteilen. Auf Unterschieden in der Löslichkeit und in der Molekülstruktur beruht die Einteilung in globuläre P. und fibrilläre P. Die globulären P. (Sphäroproteine) sind in Wasser und verd. Salzlösungen löslich. Sie sind kugelförmig gebaut (Rotationsellipsoide). Die definierte Faltung der Sekundärstrukturelemente der Polypeptidketten beruht im wesentlichen auf hydrophoben Wechselwirkungen zwischen unpolaren Aminosäureseitenketten und anderen nicht kovalenten Bindungen. Die gute Löslichkeit beruht auf den an der Moleküloberfläche lokalisierten, geladenen, hydrophilen Aminosäureresten, die – umgeben von einer Hydrathülle – für einen engen Kontakt mit dem Lösungsmittel sorgen. Zu den globulären P. gehören alle Enzyme und die meisten anderen biologisch aktiven P., z. B. die Hämoglobine. Die fibrillären P. (Linearproteine, Faserproteine) sind praktisch in Wasser und Salzlösungen unlöslich. Die Polypeptidketten sind hier parallel zueinander geordnet und bilden in Form langer Fasern unter anderem die Strukturelemente des Bindegewebes. Wichtige Vertreter sind die Kollagene, Keratine und Elastine. Nach den Bestandteilen unterscheidet man einfache P., die nur aus proteinogenen Aminosäuren aufgebaut sind, und Proteide (zusammengesetzte P., konjugierte P.), die neben dem Proteinanteil eine meist chemisch gebundene Nichtproteinkomponente enthalten. Einfache P. sind die Albumine, Globine, Globuline, Gluteline, Histone, Prolamine, Protamine, die sämtlich globuläre P. sind, und die fibrillären Skleroproteine.
Aufbau und Struktur. Am Aufbau der P. sind 21 unterschiedliche Aminosäuren (auch proteinogene Aminosäuren genannt) beteiligt. Sie sind durch Peptidbindungen miteinander verknüpft, wobei die Reihenfolge (Sequenz) der Bausteine genetisch determiniert ist.
Zur Charakterisierung des strukturellen Aufbaus der P. wurden die auch für andere Biopolymere gültigen Bezeichnungen Primär-, Sekundär-, Tertiär-, Quartärstruktur eingeführt. Unter Primärstruktur versteht man die Anzahl und Sequenz der miteinander verknüpften Aminosäurebausteine, die durch Sequenzanalyse ermittelt werden kann. P. enthalten in der Regel mehr als 100 Aminosäuren in einer Polypeptidkette. Zur Sequenzanalyse wird die Kette stufenweise vom N-terminalen oder C-terminalen Ende her abgebaut, und es wird eine Identifizierung der abgespaltenen Aminosäurereste vorgenommen. Das wichtigste chem. Abbauverfahren ist der inzwischen vollautomatisierte Edman-Abbau. Gewöhnlich werden die Polypeptidketten vor der Sequenzanalyse durch chem. oder enzymatische Partialhydrolyse in kleinere Bruchstücke zerlegt. Bei der enzymatischen Spaltung werden vor allem die spezifischen Proteasen, das bekannteste chem. Spaltreagens ist Bromcyan, das spezifisch Peptidbindungen spaltet, an denen die Carboxygruppe von Methionin beteiligt ist.
Die Sekundärstruktur beschreibt die durch die Ausbildung von Wasserstoffbrücken zwischen den Carbonylsauerstoff- und Amidstickstoffatomen gegenüberliegender Peptidgruppen auftretenden spezifischen Faltungen der Polypeptidkette. Entstehen die H-Brücken innerhalb einer Kette, so bildet sich eine Schraubenstruktur (α-Helix, Abb. 1 a); liegen intermolekulare H-Brücken vor, so entsteht die Faltblattstruktur (β-Struktur, Abb. 1 b). Ein P. mit einem sehr hohen Gehalt an α-Helix ist das Myoglobin (> 75 %), eine vollständige Faltblattstruktur findet sich im Seidenfibroin. Die α-Helix des Haarkeratins kann durch Dehnung im feuchten Zustand reversibel in eine β-Struktur umgewandelt werden. Zur Untersuchung der Sekundärstruktur von P. werden Messungen der optischen Rotationsdispersion (ORD) und des Cirkulardichroismus (CD) herangezogen (chiroptische Methoden).

Proteine. Abb.1: Sekundärstruktur von Proteinen: (a) α-Helix, (b) antiparallele Faltblattstruktur.

Proteine. Abb. 2: Tertiärstruktur der Ribonuclease A nach Kartha.
Unter Tertiärstruktur wird die durch intramolekulare Wechselwirkung der Seitenkettenfunktionen verursachte räumliche Faltung der Polypeptidkette verstanden (Abb. 2). In dieser Struktur, die insbesondere durch Disulfidbrücken stabilisiert wird, ist die Lage reaktiver Aminosäurereste z. B. im aktiven Zentrum von Enzymen oder in der Antigenbindungsstelle von Antikörpern bis in den atomaren Bereich fixiert. Die Aufklärung der Tertiärstruktur erfolgt durch Röntgenkristallstrukturanalyse isomorph kristallisierter Schweratomderivate der P. Die erhaltenen Beugungsdiagramme und die daraus gewonnene Elektronendichteverteilungskurven geben Aufschluß über die Lage der Aminosäurereste, bei hoher Auflösung (0,15 nm) auch über die Atomabstände im Proteinmolekül.
Durch intermolekulare Wechselwirkungen zwischen zwei oder mehreren identischen oder verschiedenen Polypeptidketten kommt es infolge Assoziation der Ketten zu stabilen oligomeren P. Diese geordneten Assoziate stellen die Quartärstruktur eines P. dar. Der größte Teil der quartären P. ist aus nicht kovalent verbundenen Untereinheiten aufgebaut. Die Untersuchung von Quartärstrukturen erfolgt auf elektronenmikroskopischem Wege oder durch Röntgenstrukturanalyse (Abb. Hämoglobin). Es gilt allgemein, daß die P. mit Quartärstruktur in bezug auf die Flexibilität ihrer Gestalt und Aktivität den physiologischen Bedingungen optimal angepaßt sind.
Eigenschaften. Alle P. haben eine hohe relative Molekülmasse Mr, die unter anderem durch Bestimmung der Diffusions- und Sedimentationsgeschwindigkeit in der Ultrazentrifuge, durch Messung der Licht- und Röntgenkleinwinkelstreuung, durch osmotische und elektrophoretische Messungen sowie durch Bestimmung der Wanderungsgeschwindigkeit in Dextran- oder Polyacrylamidgelen ermittelt werden kann. Die Mr der Einkettenproteine liegen zwischen 10000 und 100000, die der Mehrkettenproteine im Bereich von etwa 50000 und mehreren Millionen. Entsprechend ihrer Molekülgröße und -gestalt (Abmessungen zwischen 2 und 100 nm) gehören die P. zu den Kolloiden. Sie dialysieren nicht, bilden keine echten Lösungen, zeigen den Tyndall-Effekt und weisen eine relativ hohe Viskosität auf. Infolge der großen Anzahl ionisierter Gruppen im Molekül haben die P. hohe Dipolmomente. Besonders charakteristisch ist die Ampholytnatur der P. Sie beruht auf der gleichzeitigen Anwesenheit freier saurer und basischer Gruppen im Proteinmolekül. Der Ladungszustand des Gesamtmoleküls hängt vom pH-Wert der Lösung ab. Im stark sauren Medium liegen Polykationen, im stark basischen Polyanionen vor. Durch die resultierende positive bzw. negative Überschußladung nehmen Hydratation und Löslichkeit zu. Dabei ist für die Hydratation allein die Absolutladung entscheidend. Der Ladungssinn ist für das elektrophoretische Verhalten, d. h. für die Wanderungsrichtung im elektrischen Feld, verantwortlich. Am isoelektrischen Punkt haben die P. keine Nettoüberschußladung. In der hier vorliegenden Zwitterionenform erreichen Löslichkeit und Hydratation ein Minimum. Die Ampholytnatur der P. ist von entscheidender Bedeutung für ihre Pufferwirkung in biologischen Systemen. Aufgrund der Hydratation sind die globulären P. in der Lage, hydrophobe Substanzen einzuschließen und vor Ausflockung zu schützen. Diese Schutzkolloidfunktion ist für die Stabilisierung von Körperflüssigkeiten wichtig. Durch den Zusatz schwach- oder nichtpolarer Lösungsmittel (z. B. Alkohol oder Aceton) kommt es ebenso wie durch hohe Neutralsalzkonzentrationen zur Entfernung der Hydrathülle und damit zur Ausflockung (Aussalzung) der P. Werden P. auf Temperaturen über 60 °C erhitzt, so entstehen tiefgreifende strukturelle Veränderungen, die gleichzeitig zum Verlust oder zur Beeinträchtigung der biologischen Aktivität der betreffenden P. führen. Diese Denaturierung beruht auf der Zerstörung der Tertiär- und Quartärstruktur der P. Sie kann außer durch Erhitzen auch durch UV- und Röntgenbestrahlung, durch extrem saure oder alkalische Behandlung, durch Einwirkung von Netzmitteln, z. B. von 1%iger Natriumdodecylsulfatlösung, oder durch wasserstoffbrückenlösende Reagenzien wie 8 M Harnstoff- und 6 M Guanidinhydrochloridlösung erfolgen. Wird die Denaturierung in Gegenwart von Reduktionsmitteln vorgenommen, so werden außer den nichtkovalenten Bindungen auch die Disulfidbindungen gespalten. Ist die Denaturierung reversibel, so kann der native Zustand des P. wiederhergestellt werden (Renaturierung). Bei irreversibler Denaturierung, z. B. bei der Hitzedenaturierung des Ovalbumins (Kochen des Hühnereis), kommt es zur Ausbildung ungeordneter Gerüstkonformationen, die auch als statistische Knäuel (random coil) bezeichnet werden.
Nachweis und Bestimmung. P. können qualitativ durch Fällungsreaktionen mit Trichloressig-, Pikrin- oder Perchlorsäure, durch Schwermetall-Ionen (Cu-, Fe-, Zn- und Pb-Salze) oder durch spezifische Farbreaktionen nachgewiesen werden. Bei der Xanthoproteinreaktion z. B. entsteht eine Gelbfärbung beim Versetzen mit konz. Salpetersäure, bei der Biuretreaktion eine Purpurviolettfärbung durch Zusatz von Kupfersulfat zur stark alkalischen Proteinlösung und bei der Pauly-Reaktion eine Rotfärbung durch Behandlung der alkalischen Lösung mit Diazobenzensulfonsäure. Zur quantitativen P.-Bestimmung dient vor allem die Lowry-Methode. Hier wird ein mit dem P. gebildeter Kupferphosphomolybdänsäurekomplex (Absorptionsmaximum 750 nm) kolorimetrisch bestimmt und Human- oder Rinderserumalbumin als Eichsubstanz verwendet.
Beim klassischen Kjeldahl-Verfahren wird die Analysenprobe durch Kochen in konz. Schwefelsäure aufgeschlossen, wobei sich eine dem Stickstofffgehalt des P. äquivalente Menge Ammoniumsulfat bildet. Das daraus durch Alkalilauge freigesetzte Ammoniak wird acidimetrisch bestimmt. Eine direkte und schnelle Bestimmung von P. ist durch die Messung der UV-Absorption bei 280 nm möglich. Sie beruht auf der Anwesenheit von aromatischen Aminosäureresten (Tyr, Trp) in den meisten P.
Isolierung und Reindarstellung. Während die Isolierung der in hohen Konzentrationen vorkommenden globulären P., z. B. von Hämoglobin aus Erythrocyten, von Casein aus Milch und von Ovalbumin aus dem Eiklar, sowie die Isolierung der unlöslichen fibrillären Strukturproteine keine besonderen Schwierigkeiten bereitet, erfordert die Gewinnung der nur in geringen Mengen auftretenden P. meist eine aufwendige Abtrennung von Begleitstoffen wie Kohlenhydraten, Lipiden, Nucleinsäuren und anderen Biomolekülen. Im ersten Schritt wird das biologische Material aufgeschlossen. Nach mechanischem Zerkleinern durch Homogenisatoren, Ultraschall, Schütteln mit Glasperlen, Zermörsern des in Aluminiumoxidkörnern eingefrorenen Gewebes, durch Detergensbehandlung u. a. erhält man ein Zellhomogenisat, aus dem die P. mit Salzlösungen, Glycerin, verd. Säuren oder anderen Extraktionsmitteln herausgelöst werden. Im zweiten Schritt erfolgt eine Vortrennung der P. durch fraktionierte Ammoniumsulfatfällung oder durch Lösungsmittelfraktionierung nach Cohn, anschließend eine weitere Reinigung durch Gelfiltration, Ionenaustausch- oder Adsorptionschromatographie, gegebenenfalls auch durch präparative Elektrophorese, durch Elektrofokussierung, durch Affinitätschromatographie oder durch Ionenfiltrationschromatographie, d. i. eine Kombination von Ionenaustausch- und Gelchromatographie.
Biosynthese. Hierbei werden freie Aminosäuren des Zellbereichs in genetisch determinierter Reihenfolge peptidartig miteinander verknüpft. An der Synthese sind als Matrize dienende Messenger-RNA (mRNA), die die Aminosäuren esterartig bindenden und transportierenden Transfer-RNA (tRNA), die in den Ribosomenuntereinheiten lokalisierten ribosomalen rRNA, eine Reihe spezifischer Ribosomenproteine und -enzyme, verschiedene niedermolekulare Cofaktoren sowie ATP (Adenosin-5'-triphosphat) und GTP (Guanosin-5'-triphosphat) als Energielieferanten kooperativ beteiligt. Die Biosynthese beginnt im Zellkern mit der Kopie einer mRNA, wobei eine entsprechende chromosomale DNA als Matrize fungiert (Transcription). Die gebildete mRNA wandert in das Cytoplasma und dient hier, an das Ribosom einer Polysomenkette des endoplasmatischen Reticulums gebunden, als Matrize für den Aufbau der Polypeptidketten (Translation). Die Ribosomen bestehen aus einer großen und kleinen Untereinheit mit spezifischen Bindungsstellen für die anzubauenden Aminosäuren und die wachsende Peptidkette. Die mRNA bewegt sich in einer "Spurrinne" zwischen den beiden Untereinheiten (Abb. 3).
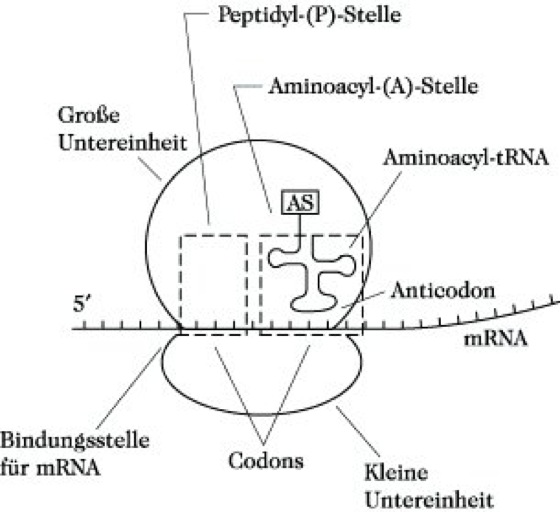
Proteine. Abb. 3: Schematische Darstellung der Ribosomenstruktur von Escherichia coli nach Lehninger (AS Aminosäure).
Die für die Peptidverknüpfung erforderliche Aktivierung der Aminosäuren erfolgt durch kovalente Bindung an hochspezifische tRNA-Moleküle. Es entstehen "aktivierte Ester" mit hohem Gruppenübertragungspotential, die als Aminoacyl-tRNA an die entsprechende Bindungsstelle transportiert und angelagert werden. Als Initiatoraminosäure fungiert bei der Synthese in E. coli das durch das Nucleotidtriplett AUG codierte Methionin, das als Formylmethionyl-tRNA gebunden wird. Die Knüpfung der ersten Peptidbindung erfolgt nach Anlagerung der zweiten Aminoacyl-tRNA durch nucleophilen Angriff der Aminogruppe auf die Esterbindung der benachbarten Formyl-Met-tRNA. Durch Weitertransport der entstandenen Dipeptidyl-tRNA um die Länge eines Codetripletts wird der Akzeptorort für die nachfolgende Anknüpfung freigegebenen (Abb. 4, S 113.). Stop-Codons geben das Signal für das Ende einer Synthese. Andere Signalsequenzen regulieren spezifische Synthesewege, z. B. die lokalspezifische Bildung von P., die die Zelle über Kanalsysteme des endoplasmatischen Reticulums verlassen sollen (Sekretproteine). Die Signalcodons folgen unmittelbar auf das Startcodon AUG. Der größte Teil der synthetisierten P. verläßt das Ribosom in biologisch aktiver Form. Die Abtrennung des Startmethionins und der Signalpeptide erfolgt auf enzymatischem Wege. Eine Reihe von P. wird im Anschluß an die Synthese durch Hydroxylierung, Methylierung, Prenylierung oder Phosphorylierung modifiziert ("Processing") oder an Nichtproteinkomponenten gebunden.
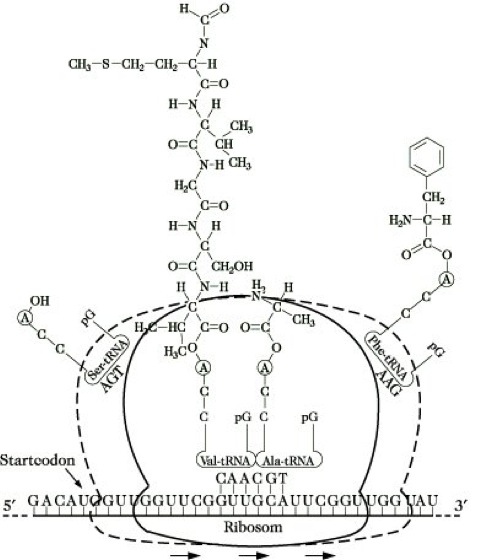
Proteine. Abb. 4: Schematische Darstellung der Translation.
Die Regulation der mit außerordentlicher Präzision und hoher Geschwindigkeit (bis zu 100 Peptidbindungen je Sekunde werden geknüpft!) verlaufenden Biosynthese von P. erfolgt auf der Transcriptions- und Translationsebene. Auf der spezifischen Hemmung von Einzelschritten beruht die Wirkung einer Reihe von Antibiotika: Chloramphenicol z. B. stört die Translation, indem es Peptidylübertragungsreaktionen blockiert, Rifamycin stört die Transcription durch spezifische Hemmung von RNA-Polymerasen.
P. als Nahrungsmittel. Die P. sind im Gegensatz zu den austauschbaren Kohlenhydraten und Fetten essentieller Grundbestandteil der menschlichen und tierischen Nahrung. Die hohe Wertigkeit der tierischen P., z. B. des Hühnereis, des Rindfleischs und der Milch, beruht darauf, daß sie die essentiellen Aminosäuren sowohl in ausreichender Menge als auch in einem für den menschlichen Bedarf günstigen Verhältnis zueinander enthalten. Die pflanzlichen P. enthalten gewöhnlich zu wenig Lysin, Methionin, Tryptophan oder Threonin. Das Defizit kann durch Zusatz der limitierenden Aminosäuren oder durch günstige Proteinkombinationen ausgeglichen werden.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.